3.2 Datos limpios
Una vez que importamos datos a R es conveniente limpiarlos, esto implica almacenarlos de una manera consisistente que nos permita enfocarnos en responder preguntas de los datos en lugar de estar luchando con los datos.
Datos limpios son datos que facilitan las tareas del análisis de datos:
Visualización: Resúmenes de datos usando gráficas, análisis exploratorio, o presentación de resultados.
Manipulación: Manipulación de variables como agregar, filtrar, reordenar, transformar.
Modelación: Ajustar modelos es sencillo si los datos están en la forma correcta.
Los principios de los datos limpios (Wickham 2014) proveen una manera estándar de organizar la información:
- Cada columna es una variable.
- Cada renglón es una observación .
- Cada celda es un único valor.
Vale la pena notar que los principios de los datos limpios se pueden ver como teoría de algebra relacional para estadísticos, estós principios junto con cada tipo de unidad observacional forma una tabla equivalen a la tercera forma normal de Codd con enfoque en una sola tabla de datos en lugar de muchas conectadas en bases de datos relacionales.
Veamos un ejemplo:
La mayor parte de las bases de datos en estadística tienen forma rectangular, ¿cuántas variables tiene la siguiente tabla?
| tratamientoA | tratamientoB | |
|---|---|---|
| Juan Aguirre | - | 2 |
| Ana Bernal | 16 | 11 |
| José López | 3 | 1 |
La tabla anterior también se puede estructurar de la siguiente manera:
| Juan Aguirre | Ana Bernal | José López | |
|---|---|---|---|
| tratamientoA | - | 16 | 3 |
| tratamientoB | 2 | 11 | 1 |
Si vemos los principios (cada variable forma una columna, cada observación forma un renglón, cada tipo de unidad observacional forma una tabla), ¿las tablas anteriores cumplen los principios?
Para responder la pregunta identifiquemos primero cuáles son las variables y cuáles las observaciones de esta pequeña base. Las variables son: persona/nombre, tratamiento y resultado. Entonces, siguiendo los principios de datos limpios obtenemos la siguiente estructura:
| nombre | tratamiento | resultado |
|---|---|---|
| Juan Aguirre | a | - |
| Ana Bernal | a | 16 |
| José López | a | 3 |
| Juan Aguirre | b | 2 |
| Ana Bernal | b | 11 |
| José López | b | 1 |
Limpieza bases de datos
Los principios de los datos limpios parecen obvios pero la mayor parte de los datos no los cumplen debido a:
- La mayor parte de la gente no está familiarizada con los principios y es
difícil derivarlos por uno mismo.
- Los datos suelen estar organizados para facilitar otros aspectos que no son análisis, por ejemplo, la captura.
Algunos de los problemas más comunes en las bases de datos que no están limpias son:
- Los encabezados de las columnas son valores y no nombres de variables.
- Más de una variable por columna.
- Las variables están organizadas tanto en filas como en columnas.
- Más de un tipo de observación en una tabla.
- Una misma unidad observacional está almacenada en múltiples tablas.
La mayor parte de estos problemas se pueden arreglar con pocas herramientas,
a continuación veremos como limpiar datos usando 2 funciones del paquete
tidyr:
pivot_longer(): recibe múltiples columnas y las convierte en pares de valores y nombres de tal manera que alarga los datos.
pivot_wider(): el opuesto apivot_longer()recibe columnas que separa haciendo los datos más anchos.
Repasaremos los problemas más comunes que se encuentran en conjuntos de datos sucios y mostraremos como se puede manipular la tabla de datos (usando las funciones de pivoteo) con el fin de estructurarla para que cumpla los principios de datos limpios.
Nota: Quizá has visto código de tidyr usando las funciones gather() y
spread(), estas son versiones anteriores a las funciones de pivoteo, sin
embargo, se les seguirá dando mantenimiento puesto que son muy populares, aquí puedes encontrar
una versión de las notas usando que utilizan gather() y spread().
Los encabezados de las columanas son valores
Usaremos ejemplos para entender los conceptos más facilmente. Comenzaremos con una tabla de datos que contiene las mediciones de partículas suspendidas PM2.5 de la red automática de monitoreo atmosférico (RAMA) para los primeros meses del 2019.
library(tidyverse)
library(estcomp)
pm25_2019
#> # A tibble: 5,088 x 26
#> date hour AJM AJU BJU CAM CCA COY FAR GAM HGM INN
#> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <lgl> <lgl> <dbl> <dbl> <dbl>
#> 1 2019-01-01 1 19 35 62 90 66 NA NA NA 56 22
#> 2 2019-01-01 2 17 24 88 104 84 NA NA NA 61 14
#> 3 2019-01-01 3 14 20 107 140 95 NA NA NA 74 8
#> 4 2019-01-01 4 6 15 101 162 97 NA NA NA 90 9
#> 5 2019-01-01 5 4 8 121 133 88 NA NA NA 90 3
#> 6 2019-01-01 6 7 7 93 106 77 NA NA NA 106 3
#> 7 2019-01-01 7 12 8 84 98 51 NA NA NA 115 NA
#> 8 2019-01-01 8 15 7 101 82 39 NA NA NA 90 NA
#> 9 2019-01-01 9 24 3 89 54 26 NA NA NA 90 NA
#> 10 2019-01-01 10 24 NA 88 76 26 NA NA NA 99 2
#> # … with 5,078 more rows, and 14 more variables: MER <dbl>, MGH <dbl>,
#> # MON <dbl>, MPA <lgl>, NEZ <dbl>, PED <dbl>, SAC <lgl>, SAG <dbl>,
#> # SFE <dbl>, SJA <lgl>, TLA <dbl>, UAX <dbl>, UIZ <dbl>, XAL <dbl>¿Cuáles son las variables en estos datos?
Esta base de datos tiene 4 variables: fecha, hora, estación y medición (en microgramos por metro cúbico \(\mu g/m^3\)).
Al alargar los datos desaparecerán las columnas que se agrupan y darán
lugar a dos nuevas columnas: la correspondiente a estación y la
correspondiente a medición. Entonces, usamos la función pivot_longer() que
recibe los argumentos:
- data:
data.frameque vamos a pivotear, alargar.
- cols: columnas que vamos a pivotear (apilar), la
notación para seleccionarlas es
tidyselect, la misma que usamos conselect()endplyr. - names_to: nombre (
string: en comillas “”) de la nueva columna que almacenará los nombres de las columnas en los datos.
- values_to: nombre (
string: en comillas “”) de la nueva columna que almacenará los valores en los datos.
pm25_2019_tidy <- pivot_longer(pm25_2019, cols = AJM:XAL, names_to = "station",
values_to = "measurement")
pm25_2019_tidy
#> # A tibble: 122,112 x 4
#> date hour station measurement
#> <date> <dbl> <chr> <dbl>
#> 1 2019-01-01 1 AJM 19
#> 2 2019-01-01 1 AJU 35
#> 3 2019-01-01 1 BJU 62
#> 4 2019-01-01 1 CAM 90
#> 5 2019-01-01 1 CCA 66
#> 6 2019-01-01 1 COY NA
#> 7 2019-01-01 1 FAR NA
#> 8 2019-01-01 1 GAM NA
#> 9 2019-01-01 1 HGM 56
#> 10 2019-01-01 1 INN 22
#> # … with 122,102 more rowsObservemos que en la tabla original teníamos bajo la columna AJM, en el renglón correspondiente a 2019-01-01 hora 1 un valor de 19, y podemos ver que este valor en la tabla larga se almacena bajo la columna measurement y corresponde a la estación AJM.
La nueva estructura de la base de datos nos permite, por ejemplo, hacer fácilmente una gráfica donde podemos comparar las diferencias en las frecuencias.
pm25_2019_tidy %>%
mutate(
missing = is.na(measurement),
station = reorder(station, missing, sum)
) %>%
ggplot(aes(x = date, y = hour, fill = is.na(measurement))) +
geom_raster(alpha = 0.8) +
facet_wrap(~ station) +
scale_fill_manual("faltante",
values = c("TRUE" = "salmon", "FALSE" = "gray"))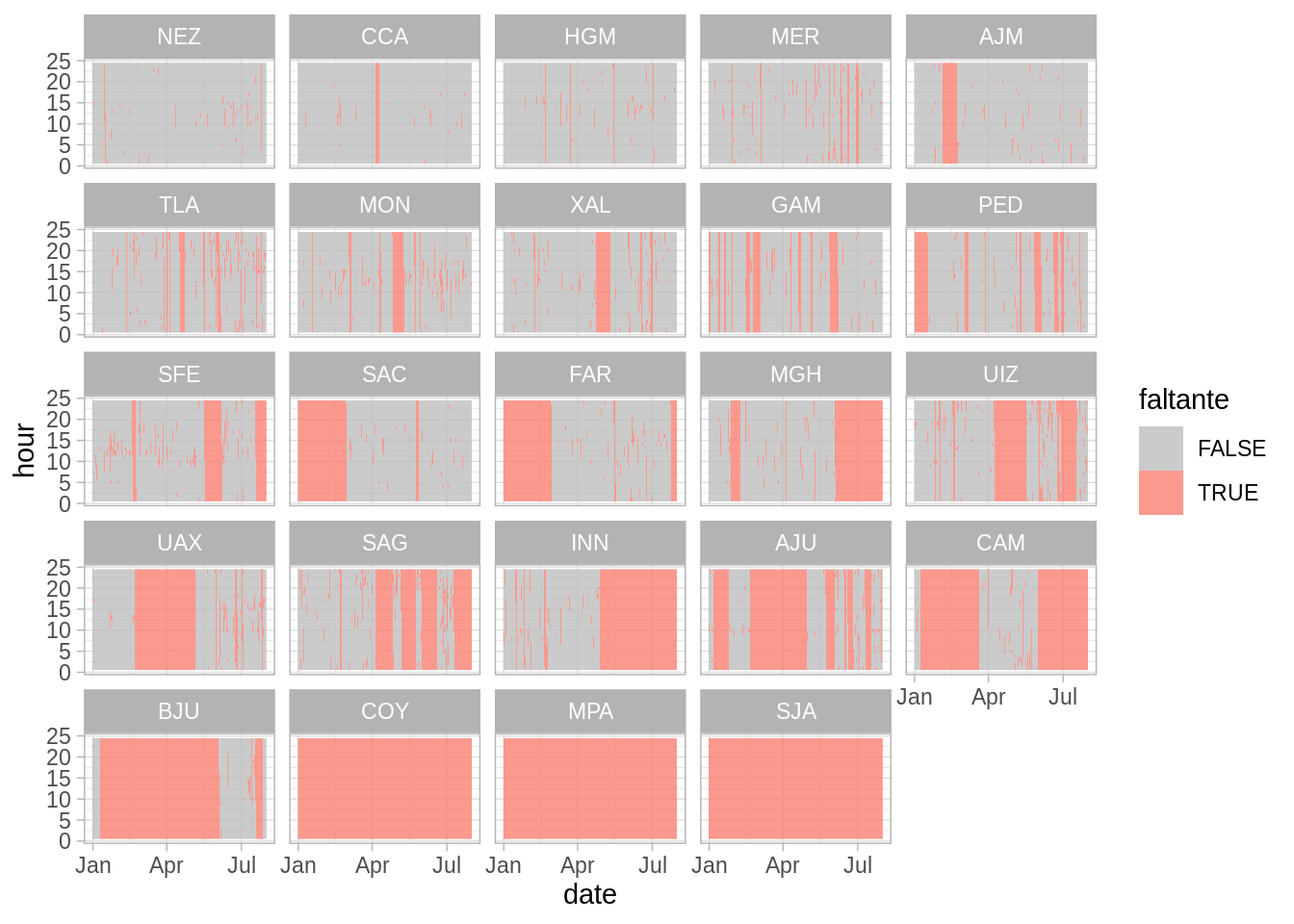
Otro ejemplo, veamos los datos df_edu, ¿cuántas variables tenemos?
df_edu
#> # A tibble: 7,371 x 16
#> state_code municipio_code region state_name state_abbr municipio_name sex
#> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 01 001 01001 Aguascali… AGS Aguascalientes Total
#> 2 01 001 01001 Aguascali… AGS Aguascalientes Homb…
#> 3 01 001 01001 Aguascali… AGS Aguascalientes Muje…
#> 4 01 002 01002 Aguascali… AGS Asientos Total
#> 5 01 002 01002 Aguascali… AGS Asientos Homb…
#> 6 01 002 01002 Aguascali… AGS Asientos Muje…
#> 7 01 003 01003 Aguascali… AGS Calvillo Total
#> 8 01 003 01003 Aguascali… AGS Calvillo Homb…
#> 9 01 003 01003 Aguascali… AGS Calvillo Muje…
#> 10 01 004 01004 Aguascali… AGS Cosío Total
#> # … with 7,361 more rows, and 9 more variables: pop_15 <dbl>, no_school <dbl>,
#> # preschool <dbl>, elementary <dbl>, secondary <dbl>, highschool <dbl>,
#> # higher_edu <dbl>, other <dbl>, schoolyrs <dbl>Notemos que el nivel de escolaridad esta guardado en 6 columnas (preschool, elementary, …, other), este tipo de almacenamiento no es limpio aunque puede ser útil al momento de ingresar la información o para presentarla.
Para tener datos limpios apilamos los niveles de escolaridad de manera que sea una sola columna (nuevamente alargamos los datos):
df_edu_tidy <- pivot_longer(data = df_edu, cols = preschool:other,
names_to = "grade", values_to = "percent", values_drop_na = TRUE)
glimpse(df_edu_tidy)
#> Observations: 44,226
#> Variables: 12
#> $ state_code <chr> "01", "01", "01", "01", "01", "01", "01", "01", "01", …
#> $ municipio_code <chr> "001", "001", "001", "001", "001", "001", "001", "001"…
#> $ region <chr> "01001", "01001", "01001", "01001", "01001", "01001", …
#> $ state_name <chr> "Aguascalientes", "Aguascalientes", "Aguascalientes", …
#> $ state_abbr <chr> "AGS", "AGS", "AGS", "AGS", "AGS", "AGS", "AGS", "AGS"…
#> $ municipio_name <chr> "Aguascalientes", "Aguascalientes", "Aguascalientes", …
#> $ sex <chr> "Total", "Total", "Total", "Total", "Total", "Total", …
#> $ pop_15 <dbl> 631064, 631064, 631064, 631064, 631064, 631064, 301714…
#> $ no_school <dbl> 2.662329, 2.662329, 2.662329, 2.662329, 2.662329, 2.66…
#> $ schoolyrs <dbl> 10.211152, 10.211152, 10.211152, 10.211152, 10.211152,…
#> $ grade <chr> "preschool", "elementary", "secondary", "highschool", …
#> $ percent <dbl> 0.17335801, 20.15247265, 29.31144860, 23.31823714, 24.…El parámetro values_drop_na = TRUE se utiliza para eliminar los
renglones con valores faltantes en la columna de porcentaje, esto es, eliminamos
aquellas observaciones que tenían NA en la columnas de nivel de escolaridad de
la tabla ancha. En este caso optamos por que los faltantes sean implícitos, la
conveniencia de tenerlos implícitos/explícitos dependerá de la aplicación.
Con los datos limpios es facil hacer manipulaciones y grfiacs, ¿cómo habrían hecho la siguiente gráfica antes de la limpieza?
df_edu_cdmx <- df_edu_tidy %>%
filter(state_abbr == "CDMX", sex != "Total", grade != "other") %>%
mutate(municipio_name = reorder(municipio_name, percent, last))
ggplot(df_edu_cdmx, aes(x = grade,
y = percent, group = sex, color = sex)) +
geom_path() +
facet_wrap(~municipio_name) +
theme(axis.text.x = element_text(angle = 60, hjust = 1)) +
scale_x_discrete(limits = c("preschool", "elementary",
"secondary", "highschool", "higher_edu"))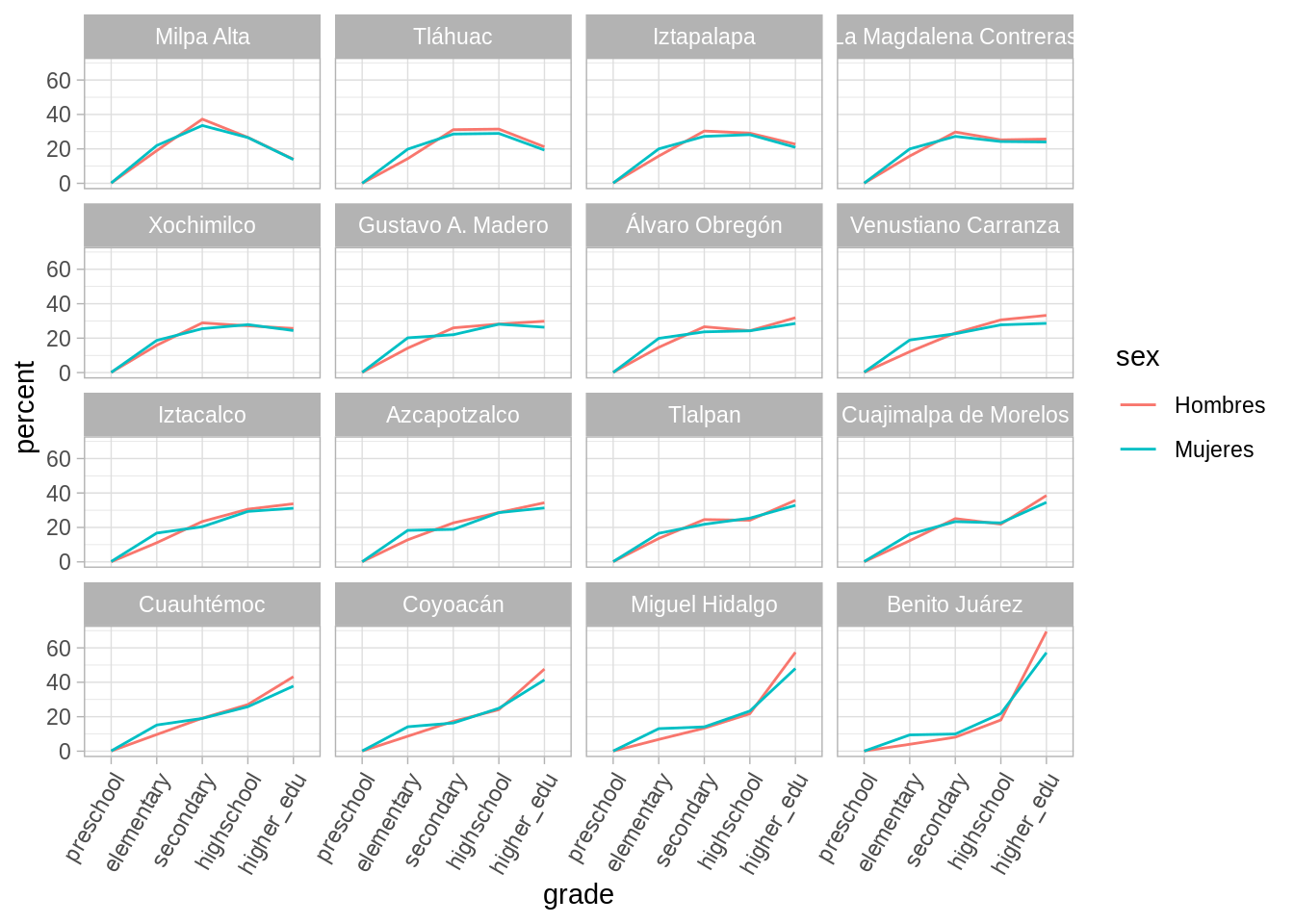
Una columna asociada a más de una variable
Utilizaremos un subconjunto de los datos de la prueba ENLACE a nivel primaria, la prueba ENLACE evaluaba a todos los alumnos de tercero a sexto de primaria y a los alumnos de secundaria del país en 3 áreas: español, matemáticas y formación cívica y ética.
data("enlacep_2013")
enlacep_sub_2013 <- enlacep_2013 %>%
select(CVE_ENT:PUNT_FCE_6) %>%
sample_n(1000)
glimpse(enlacep_sub_2013)
#> Observations: 1,000
#> Variables: 22
#> $ CVE_ENT <chr> "21", "05", "05", "09", "21", "21", "15", "08", "19", "14"…
#> $ NOM_ENT <chr> "PUEBLA", "COAHUILA", "COAHUILA", "DISTRITO FEDERAL", "PUE…
#> $ CCT <chr> "21KPB0099S", "05DPR1382W", "05DPR0276F", "09PPR1529Z", "2…
#> $ TURNO <chr> "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO"…
#> $ ESCUELA <chr> "PRIMARIA COMUNITARIA INDIGENA CALAPANA", "DR. JOSE IDU\u0…
#> $ TIPO <chr> "CONAFE", "GENERAL", "GENERAL", "PARTICULAR", "INDêGENA", …
#> $ CVE_MUN <chr> "123", "024", "035", "007", "076", "208", "058", "040", "0…
#> $ NOM_MUN <chr> "SAN FELIPE TEPATLAN", "PARRAS", "TORREON", "IZTAPALAPA", …
#> $ CVE_LOC <chr> "0017", "0112", "0001", "0091", "0154", "0016", "0001", "0…
#> $ NOM_LOC <chr> "CALAPANA", "SOMBRERETILLO", "TORREON", "PARAJE SAN JUAN",…
#> $ PUNT_ESP_3 <dbl> 591, 450, 564, 659, 540, 627, 520, 534, 474, 559, 472, 521…
#> $ PUNT_MAT_3 <dbl> 539, 481, 632, 702, 572, 666, 533, 561, 511, 612, 513, 579…
#> $ PUNT_FCE_3 <dbl> 503, 420, 542, 581, 511, 550, 468, 496, 428, 518, 399, 484…
#> $ PUNT_ESP_4 <dbl> 457, 453, 536, 580, 576, 663, 546, 564, 512, 615, 435, 613…
#> $ PUNT_MAT_4 <dbl> 430, 436, 575, 570, 628, 683, 590, 568, 566, 681, 426, 706…
#> $ PUNT_FCE_4 <dbl> 463, 439, 452, 513, 503, 618, 494, 551, 444, 498, 397, 593…
#> $ PUNT_ESP_5 <dbl> 475, 575, 608, 610, 575, 530, 504, 485, 420, 586, 416, 738…
#> $ PUNT_MAT_5 <dbl> 528, 587, 620, 576, 662, 507, 512, 490, 448, 704, 483, 823…
#> $ PUNT_FCE_5 <dbl> 477, 498, 569, 532, 537, 473, 476, 466, 408, 550, 388, 675…
#> $ PUNT_ESP_6 <dbl> 431, 521, 455, 568, 580, 505, 507, 466, 475, 589, 414, 607…
#> $ PUNT_MAT_6 <dbl> 501, 435, 539, 571, 659, 607, 506, 523, 489, 698, 470, 635…
#> $ PUNT_FCE_6 <dbl> 374, 436, 375, 490, 568, 479, 474, 387, 445, 585, 447, 511… ¿Cuántas variables tiene este subconjunto de los datos?
¿Cuántas variables tiene este subconjunto de los datos?
De manera similar a los ejemplos anteriores, utiliza la función
pivot_longerpara apilar las columnas correspondientes a área-grado.Piensa en como podemos separar la “variable” área-grado en dos columnas.
Ahora separaremos las variables área y grado de la columna AREA_GRADO,
para ello debemos pasar a la función separate(), esta recibe como parámetros:
el nombre de la base de datos,
el nombre de la variable que deseamos separar en más de una,
la posición de donde deseamos “cortar” (hay más opciones para especificar como separar, ver
?separate). El default es separar valores en todos los lugares que encuentre un caracter que no es alfanumérico (espacio, guión,…).
enlacep_tidy <- separate(data = enlacep_long, col = AREA_GRADO,
into = c("AREA", "GRADO"), sep = 9)
enlacep_tidy
#> # A tibble: 12,000 x 13
#> CVE_ENT NOM_ENT CCT TURNO ESCUELA TIPO CVE_MUN NOM_MUN CVE_LOC NOM_LOC
#> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
#> 1 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 2 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 3 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 4 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 5 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 6 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 7 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 8 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 9 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> 10 21 PUEBLA 21KP… MATU… PRIMAR… CONA… 123 SAN FE… 0017 CALAPA…
#> # … with 11,990 more rows, and 3 more variables: AREA <chr>, GRADO <chr>,
#> # PUNTAJE <dbl>
# creamos un mejor código de área
enlacep_tidy <- enlacep_tidy %>%
mutate(
AREA = substr(AREA, 6, 8),
GRADO = as.numeric(GRADO)
)
glimpse(enlacep_tidy)
#> Observations: 12,000
#> Variables: 13
#> $ CVE_ENT <chr> "21", "21", "21", "21", "21", "21", "21", "21", "21", "21", "…
#> $ NOM_ENT <chr> "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "…
#> $ CCT <chr> "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KP…
#> $ TURNO <chr> "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "…
#> $ ESCUELA <chr> "PRIMARIA COMUNITARIA INDIGENA CALAPANA", "PRIMARIA COMUNITAR…
#> $ TIPO <chr> "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "…
#> $ CVE_MUN <chr> "123", "123", "123", "123", "123", "123", "123", "123", "123"…
#> $ NOM_MUN <chr> "SAN FELIPE TEPATLAN", "SAN FELIPE TEPATLAN", "SAN FELIPE TEP…
#> $ CVE_LOC <chr> "0017", "0017", "0017", "0017", "0017", "0017", "0017", "0017…
#> $ NOM_LOC <chr> "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "…
#> $ AREA <chr> "ESP", "MAT", "FCE", "ESP", "MAT", "FCE", "ESP", "MAT", "FCE"…
#> $ GRADO <dbl> 3, 3, 3, 4, 4, 4, 5, 5, 5, 6, 6, 6, 3, 3, 3, 4, 4, 4, 5, 5, 5…
#> $ PUNTAJE <dbl> 591, 539, 503, 457, 430, 463, 475, 528, 477, 431, 501, 374, 4…Conforme nos habituemos a las funciones podemos sacar provecho de sus argumentos adicionales:
names_prefix: recibe una expresión regular para eliminar el texto que coincida del inicio de una variable.
pivot_longer(enlacep_sub_2013, cols = contains("PUNT"),
names_to = c("AREA_GRADO"), values_to = "PUNTAJE",
names_prefix = "PUNT_") %>%
glimpse()
#> Observations: 12,000
#> Variables: 12
#> $ CVE_ENT <chr> "21", "21", "21", "21", "21", "21", "21", "21", "21", "21"…
#> $ NOM_ENT <chr> "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA"…
#> $ CCT <chr> "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KPB0099S", "2…
#> $ TURNO <chr> "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO"…
#> $ ESCUELA <chr> "PRIMARIA COMUNITARIA INDIGENA CALAPANA", "PRIMARIA COMUNI…
#> $ TIPO <chr> "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE"…
#> $ CVE_MUN <chr> "123", "123", "123", "123", "123", "123", "123", "123", "1…
#> $ NOM_MUN <chr> "SAN FELIPE TEPATLAN", "SAN FELIPE TEPATLAN", "SAN FELIPE …
#> $ CVE_LOC <chr> "0017", "0017", "0017", "0017", "0017", "0017", "0017", "0…
#> $ NOM_LOC <chr> "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA"…
#> $ AREA_GRADO <chr> "ESP_3", "MAT_3", "FCE_3", "ESP_4", "MAT_4", "FCE_4", "ESP…
#> $ PUNTAJE <dbl> 591, 539, 503, 457, 430, 463, 475, 528, 477, 431, 501, 374…names_sep: nos permite hacer el pivoteo y separar en una misma operación, en este casonames_toconsiste en un vector con más de una entrada ynames_sepindica como separar el nombre de las columnas.
pivot_longer(enlacep_sub_2013, cols = contains("PUNT"),
names_to = c("AREA", "GRADO"), values_to = "PUNTAJE", names_prefix = "PUNT_",
names_sep = "_") %>%
glimpse()
#> Observations: 12,000
#> Variables: 13
#> $ CVE_ENT <chr> "21", "21", "21", "21", "21", "21", "21", "21", "21", "21", "…
#> $ NOM_ENT <chr> "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "…
#> $ CCT <chr> "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KP…
#> $ TURNO <chr> "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "…
#> $ ESCUELA <chr> "PRIMARIA COMUNITARIA INDIGENA CALAPANA", "PRIMARIA COMUNITAR…
#> $ TIPO <chr> "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "…
#> $ CVE_MUN <chr> "123", "123", "123", "123", "123", "123", "123", "123", "123"…
#> $ NOM_MUN <chr> "SAN FELIPE TEPATLAN", "SAN FELIPE TEPATLAN", "SAN FELIPE TEP…
#> $ CVE_LOC <chr> "0017", "0017", "0017", "0017", "0017", "0017", "0017", "0017…
#> $ NOM_LOC <chr> "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "…
#> $ AREA <chr> "ESP", "MAT", "FCE", "ESP", "MAT", "FCE", "ESP", "MAT", "FCE"…
#> $ GRADO <chr> "3", "3", "3", "4", "4", "4", "5", "5", "5", "6", "6", "6", "…
#> $ PUNTAJE <dbl> 591, 539, 503, 457, 430, 463, 475, 528, 477, 431, 501, 374, 4…names_pattern: similar anames_seppero recibe una expresión regular.
pivot_longer(enlacep_sub_2013, cols = contains("PUNT"),
names_to = c("AREA", "GRADO"), names_pattern = "PUNT_?(.*)_(.*)",
values_to = "PUNTAJE")names_ptypes, values_ptypes: permiten especificar el tipo de las nuevas columnas.
pivot_longer(enlacep_sub_2013, cols = contains("PUNT"),
names_to = c("AREA", "GRADO"), values_to = "PUNTAJE", names_prefix = "PUNT_",
names_sep = "_", names_ptypes = list(GRADO = integer())) %>%
glimpse()
#> Observations: 12,000
#> Variables: 13
#> $ CVE_ENT <chr> "21", "21", "21", "21", "21", "21", "21", "21", "21", "21", "…
#> $ NOM_ENT <chr> "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "PUEBLA", "…
#> $ CCT <chr> "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KPB0099S", "21KP…
#> $ TURNO <chr> "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "MATUTINO", "…
#> $ ESCUELA <chr> "PRIMARIA COMUNITARIA INDIGENA CALAPANA", "PRIMARIA COMUNITAR…
#> $ TIPO <chr> "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "CONAFE", "…
#> $ CVE_MUN <chr> "123", "123", "123", "123", "123", "123", "123", "123", "123"…
#> $ NOM_MUN <chr> "SAN FELIPE TEPATLAN", "SAN FELIPE TEPATLAN", "SAN FELIPE TEP…
#> $ CVE_LOC <chr> "0017", "0017", "0017", "0017", "0017", "0017", "0017", "0017…
#> $ NOM_LOC <chr> "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "CALAPANA", "…
#> $ AREA <chr> "ESP", "MAT", "FCE", "ESP", "MAT", "FCE", "ESP", "MAT", "FCE"…
#> $ GRADO <int> 3, 3, 3, 4, 4, 4, 5, 5, 5, 6, 6, 6, 3, 3, 3, 4, 4, 4, 5, 5, 5…
#> $ PUNTAJE <dbl> 591, 539, 503, 457, 430, 463, 475, 528, 477, 431, 501, 374, 4…Variables almacenadas en filas y columnas
El problema más difícil es cuando las variables están tanto en filas como en columnas, veamos una base de datos de fertilidad. ¿Cuáles son las variables en estos datos?
data("df_fertility")
df_fertility
#> # A tibble: 306 x 11
#> state size_localidad est age_15_19 age_20_24 age_25_29 age_30_34 age_35_39
#> <chr> <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 01 A… Menos de 2 50… Valor 74.2 175. 175. 102. 59.0
#> 2 01 A… Menos de 2 50… Erro… 6.71 11.0 9.35 8.05 7.29
#> 3 01 A… 2 500-14 999 … Valor 82.5 171. 140. 103. 72.0
#> 4 01 A… 2 500-14 999 … Erro… 9.79 12.5 10.4 8.76 9.08
#> 5 01 A… 15 000-49 999… Valor 72.6 146. 147. 99.0 58.7
#> 6 01 A… 15 000-49 999… Erro… 7.07 10.8 10.5 8.11 7.37
#> 7 01 A… 100 000 y más… Valor 66.3 120. 102. 84.2 53.9
#> 8 01 A… 100 000 y más… Erro… 7.57 8.66 8.98 8.59 6.61
#> 9 02 B… Menos de 2 50… Valor 89.6 158. 117. 86.0 42.0
#> 10 02 B… Menos de 2 50… Erro… 15.8 17.2 13.2 12.3 9.64
#> # … with 296 more rows, and 3 more variables: age_40_44 <dbl>, age_45_49 <dbl>,
#> # global <dbl>Estos datos tienen variables en columnas individuales (state, size_localidad), en múltiples columnas (grupo de edad, age_15_19,..) y en filas (Valor y Error estándar).
Comencemos por apilar las columnas.
fertility_long <- pivot_longer(df_fertility, cols = age_15_19:global,
names_to = "age_bracket", values_to = "value", names_prefix = "age_")
fertility_long
#> # A tibble: 2,448 x 5
#> state size_localidad est age_bracket value
#> <chr> <chr> <chr> <chr> <dbl>
#> 1 01 Aguascalientes Menos de 2 500 habitantes Valor 15_19 74.2
#> 2 01 Aguascalientes Menos de 2 500 habitantes Valor 20_24 175.
#> 3 01 Aguascalientes Menos de 2 500 habitantes Valor 25_29 175.
#> 4 01 Aguascalientes Menos de 2 500 habitantes Valor 30_34 102.
#> 5 01 Aguascalientes Menos de 2 500 habitantes Valor 35_39 59.0
#> 6 01 Aguascalientes Menos de 2 500 habitantes Valor 40_44 23.0
#> 7 01 Aguascalientes Menos de 2 500 habitantes Valor 45_49 4.49
#> 8 01 Aguascalientes Menos de 2 500 habitantes Valor global 3.06
#> 9 01 Aguascalientes Menos de 2 500 habitantes Error estándar 15_19 6.71
#> 10 01 Aguascalientes Menos de 2 500 habitantes Error estándar 20_24 11.0
#> # … with 2,438 more rowsPodemos crear algunas variables adicionales.
fertility_vars <- fertility_long %>%
mutate(
state_code = str_sub(state, 1, 2),
state_name = str_sub(state, 4)
) %>%
select(-state)
fertility_vars
#> # A tibble: 2,448 x 6
#> size_localidad est age_bracket value state_code state_name
#> <chr> <chr> <chr> <dbl> <chr> <chr>
#> 1 Menos de 2 500 habita… Valor 15_19 74.2 01 Aguascalien…
#> 2 Menos de 2 500 habita… Valor 20_24 175. 01 Aguascalien…
#> 3 Menos de 2 500 habita… Valor 25_29 175. 01 Aguascalien…
#> 4 Menos de 2 500 habita… Valor 30_34 102. 01 Aguascalien…
#> 5 Menos de 2 500 habita… Valor 35_39 59.0 01 Aguascalien…
#> 6 Menos de 2 500 habita… Valor 40_44 23.0 01 Aguascalien…
#> 7 Menos de 2 500 habita… Valor 45_49 4.49 01 Aguascalien…
#> 8 Menos de 2 500 habita… Valor global 3.06 01 Aguascalien…
#> 9 Menos de 2 500 habita… Error está… 15_19 6.71 01 Aguascalien…
#> 10 Menos de 2 500 habita… Error está… 20_24 11.0 01 Aguascalien…
#> # … with 2,438 more rowsFinalmente, la columna est no es una variable, sino que almacena el nombre
de 2 variables: Valor y Error Estándar la operación que debemos aplicar
(pivot_wider()) es el inverso de apilar (pivot_longer), sus argumentos son:
- data:
data.frameque vamos a pivotear.
- names_from: nombre o nombres de las columnas (sin comillas) de los cuáles obtendremos los nombres de las nuevas columnas.
- values_from: nombre o nombres de las columnas (sin comillas) de los cuáles obtendremos los valores que llenarán las nuevas columnas.
Y podemos mejorar los nombres de las columnas, una opción rápida es usar el paquete janitor.
fertility_tidy %>%
janitor::clean_names() %>%
glimpse()
#> Observations: 1,224
#> Variables: 6
#> $ size_localidad <chr> "Menos de 2 500 habitantes", "Menos de 2 500 habitante…
#> $ age_bracket <chr> "15_19", "20_24", "25_29", "30_34", "35_39", "40_44", …
#> $ state_code <chr> "01", "01", "01", "01", "01", "01", "01", "01", "01", …
#> $ state_name <chr> "Aguascalientes", "Aguascalientes", "Aguascalientes", …
#> $ valor <dbl> 74.2032276, 175.0281396, 174.5274362, 101.5836230, 58.…
#> $ error_estandar <dbl> 6.70671255, 11.00329648, 9.34594033, 8.04764573, 7.286…o podemos hacerlo manualmente
Ahora es inmediato no solo hacer gráficas sino también ajustar un modelo.
# ajustamos un modelo lineal donde la variable respuesta es temperatura
# máxima, y la variable explicativa es el mes
fertility_sub <- filter(fertility_tidy, age_bracket != "global")
fertility_lm <- lm(est ~ age_bracket, data = fertility_sub)
summary(fertility_lm)
#>
#> Call:
#> lm(formula = est ~ age_bracket, data = fertility_sub)
#>
#> Residuals:
#> Min 1Q Median 3Q Max
#> -55.060 -5.778 -0.383 6.874 55.133
#>
#> Coefficients:
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) 66.505 1.143 58.21 <2e-16 ***
#> age_bracket20_24 70.675 1.616 43.74 <2e-16 ***
#> age_bracket25_29 58.881 1.616 36.44 <2e-16 ***
#> age_bracket30_34 22.910 1.616 14.18 <2e-16 ***
#> age_bracket35_39 -20.312 1.616 -12.57 <2e-16 ***
#> age_bracket40_44 -53.346 1.616 -33.01 <2e-16 ***
#> age_bracket45_49 -64.797 1.616 -40.10 <2e-16 ***
#> ---
#> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#>
#> Residual standard error: 14.13 on 1064 degrees of freedom
#> Multiple R-squared: 0.922, Adjusted R-squared: 0.9215
#> F-statistic: 2096 on 6 and 1064 DF, p-value: < 2.2e-16Vale la pena notar que aunque los datos limpios facilitan las tareas de análisis, distintas funciones o tareas requieren los datos en distintos formas y saber pivotear las tablas es muy útil.
 Grafica el valor estimado de fertilidad del grupo de
edad 20-24 contra 25-29. ¿Qué transformación debes hacer? Tip: elimina la
columna que corresponde al error estándar antes de ensanchar los
datos.
Grafica el valor estimado de fertilidad del grupo de
edad 20-24 contra 25-29. ¿Qué transformación debes hacer? Tip: elimina la
columna que corresponde al error estándar antes de ensanchar los
datos.
Una misma unidad observacional está almacenada en múltiples tablas
También es común que los valores sobre una misma unidad observacional estén separados en muchas tablas o archivos, es común que estas tablas esten divididas de acuerdo a una variable, de tal manera que cada archivo representa a una persona, año o ubicación. Para juntar los archivos hacemos lo siguiente:
- Enlistamos las rutas de los archivos.
- Leemos cada archivo y agregamos una columna con el nombre del archivo.
- Combinamos las tablas en un solo data frame.
Veamos un ejemplo, descargamos la carpeta con los datos de varios contaminantes de RAMA,
ésta contiene 9 archivos de excel que almacenan información de monitoreo de contaminantes. Cada archivo contiene información de un contaminante y el nombre del archivo indica el contaminante.
Los pasos en R (usando el paquete purrr), primero creamos un vector con los
nombres de los archivos en un directorio, eligiendo aquellos que contengan las
letras “.csv”.
Después le asignamos el nombre del archivo al nombre de cada elemento del vector. Este paso se realiza para preservar los nombres de los archivos ya que estos los asignaremos a una variable mas adelante.
La función map_df itera sobre cada dirección, lee el archivo excel de dicha
dirección y los combina en un data frame.
library(readxl)
rama <- map_df(paths, read_excel, .id = "FILENAME")
# eliminamos la basura del id
rama <- rama %>%
mutate(PARAMETRO = str_remove(FILENAME, "2019") %>% str_remove(".xls")) %>%
select(PARAMETRO, FECHA:AJU)
# y apilamos para tener una columna por estación
rama_tidy <- rama %>%
gather(estacion, valor, ACO:AJU) %>%
mutate(valor = ifelse(-99, NA, valor))
rama_tidy
#> # A tibble: 1,648,512 x 5
#> PARAMETRO FECHA HORA estacion valor
#> <chr> <dttm> <dbl> <chr> <lgl>
#> 1 CO 2019-01-01 00:00:00 1 ACO NA
#> 2 CO 2019-01-01 00:00:00 2 ACO NA
#> 3 CO 2019-01-01 00:00:00 3 ACO NA
#> 4 CO 2019-01-01 00:00:00 4 ACO NA
#> 5 CO 2019-01-01 00:00:00 5 ACO NA
#> 6 CO 2019-01-01 00:00:00 6 ACO NA
#> 7 CO 2019-01-01 00:00:00 7 ACO NA
#> 8 CO 2019-01-01 00:00:00 8 ACO NA
#> 9 CO 2019-01-01 00:00:00 9 ACO NA
#> 10 CO 2019-01-01 00:00:00 10 ACO NA
#> # … with 1,648,502 more rowsOtras consideraciones
En las buenas prácticas es importante tomar en cuenta los siguientes puntos:
Incluir un encabezado con el nombre de las variables.
Los nombres de las variables deben ser entendibles (e.g. AgeAtDiagnosis es mejor que AgeDx).
En general los datos se deben guardar en un archivo por tabla.
Escribir un script con las modificaciones que se hicieron a los datos crudos (reproducibilidad).
Otros aspectos importantes en la limpieza de datos son: selección del tipo de variables (por ejemplo fechas), datos faltantes, typos y detección de valores atípicos.
Recursos adicionales
Data Transformation Cheat Sheet de RStudio.
Data Wrangling Cheat Sheet de RStudio.
Limpiar nombres de columnas, eliminar filas vacías y más, paquete janitor.
Lectura de datos tabulares con distintas estructuras, paquete tidycells.
Referencias
Wickham, Hadley. 2014. “Tidy Data.” Journal of Statistical Software, Articles 59 (10):1–23. https://doi.org/10.18637/jss.v059.i10.