3.2 Datos limpios
Una vez que importamos datos a R es conveniente limpiarlos, esto implica almacenarlos de una manera consisistente que nos permita enfocarnos en responder preguntas de los datos en lugar de estar luchando con los datos. Entonces, datos limpios son datos que facilitan las tareas del análisis de datos:
Visualización: Resúmenes de datos usando gráficas, análisis exploratorio, o presentación de resultados.
Manipulación: Manipulación de variables como agregar, filtrar, reordenar, transformar.
Modelación: Ajustar modelos es sencillo si los datos están en la forma correcta.
Los principios de datos limpios (Hadley Wickham 2014) proveen una manera estándar de organizar la información:
- Cada variable forma una columna.
- Cada observación forma un renglón.
- Cada tipo de unidad observacional forma una tabla.
Vale la pena notar que los principios de los datos limpios se pueden ver como teoría de algebra relacional para estadísticos, estós principios equivalen a la tercera forma normal de Codd con enfoque en una sola tabla de datos en lugar de muchas conectadas en bases de datos relacionales.
Veamos un ejemplo:
La mayor parte de las bases de datos en estadística tienen forma rectangular, ¿cuántas variables tiene la siguiente tabla?
| tratamientoA | tratamientoB | |
|---|---|---|
| Juan Aguirre | - | 2 |
| Ana Bernal | 16 | 11 |
| José López | 3 | 1 |
La tabla anterior también se puede estructurar de la siguiente manera:
| Juan Aguirre | Ana Bernal | José López | |
|---|---|---|---|
| tratamientoA | - | 16 | 3 |
| tratamientoB | 2 | 11 | 1 |
Si vemos los principios (cada variable forma una columna, cada observación forma un renglón, cada tipo de unidad observacional forma una tabla), ¿las tablas anteriores cumplen los principios?
Para responder la pregunta identifiquemos primero cuáles son las variables y cuáles las observaciones de esta pequeña base. Las variables son: persona/nombre, tratamiento y resultado. Entonces, siguiendo los principios de datos limpios obtenemos la siguiente estructura:
| nombre | tratamiento | resultado |
|---|---|---|
| Juan Aguirre | a | - |
| Ana Bernal | a | 16 |
| José López | a | 3 |
| Juan Aguirre | b | 2 |
| Ana Bernal | b | 11 |
| José López | b | 1 |
Limpieza bases de datos
Los principios de los datos limpios parecen obvios pero la mayor parte de los datos no los cumplen debido a:
- La mayor parte de la gente no está familiarizada con los principios y es
difícil derivarlos por uno mismo.
- Los datos suelen estar organizados para facilitar otros aspectos que no son análisis, por ejemplo, la captura.
Algunos de los problemas más comunes en las bases de datos que no están limpias son:
- Los encabezados de las columnas son valores y no nombres de variables.
- Más de una variable por columna.
- Las variables están organizadas tanto en filas como en columnas.
- Más de un tipo de observación en una tabla.
- Una misma unidad observacional está almacenada en múltiples tablas.
La mayor parte de estos problemas se pueden arreglar con pocas herramientas,
a continuación veremos como limpiar datos usando 2 funciones del paquete
tidyr:
- gather: recibe múltiples columnas y las junta en pares de valores y
nombres, convierte los datos anchos en largos.
- spread: recibe 2 columnas y las separa, haciendo los datos más anchos.
Repasaremos los problemas más comunes que se encuentran en conjuntos de datos sucios y mostraremos como se puede manipular la tabla de datos (usando las funciones gather y spread) con el fin de estructurarla para que cumpla los principios de datos limpios.
Los encabezados de las columanas son valores
Usaremos ejemplos para entender los conceptos más facilmente. La primer base de datos está basada en una encuesta de Pew Research que investiga la relación entre ingreso y afiliación religiosa.
¿Cuáles son las variables en estos datos?
library(tidyverse)
pew <- read_delim("http://stat405.had.co.nz/data/pew.txt", "\t",
escape_double = FALSE, trim_ws = TRUE)
#> Parsed with column specification:
#> cols(
#> religion = col_character(),
#> `<$10k` = col_double(),
#> `$10-20k` = col_double(),
#> `$20-30k` = col_double(),
#> `$30-40k` = col_double(),
#> `$40-50k` = col_double(),
#> `$50-75k` = col_double(),
#> `$75-100k` = col_double(),
#> `$100-150k` = col_double(),
#> `>150k` = col_double(),
#> `Don't know/refused` = col_double()
#> )
pew
#> # A tibble: 9 x 11
#> religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k`
#> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 Jewish 19 19 25 25 30 95
#> 2 Mainlin… 289 495 619 655 651 1107
#> 3 Mormon 29 40 48 51 56 112
#> 4 Muslim 6 7 9 10 9 23
#> 5 Orthodox 13 17 23 32 32 47
#> 6 Other C… 9 7 11 13 13 14
#> 7 Other F… 20 33 40 46 49 63
#> 8 Other W… 5 2 3 4 2 7
#> 9 Unaffil… 217 299 374 365 341 528
#> # ... with 4 more variables: `$75-100k` <dbl>, `$100-150k` <dbl>,
#> # `>150k` <dbl>, `Don't know/refused` <dbl>Esta base de datos tiene 3 variables: religión, ingreso y frecuencia. Para
limpiarla es necesario apilar las columnas (alargar los datos). Notemos
que al alargar los datos desapareceran las columnas que se agrupan y dan lugar a
dos nuevas columnas: la correspondiente a clave y la correspondiente a valor.
Entonces, para alargar una base de datos usamos la función gather que recibe
los argumentos:
- data: base de datos que vamos a reestructurar.
- key: nombre de la nueva variable que contiene lo que fueron los nombres
de columnas que apilamos.
- value: nombre de la variable que almacenará los valores que corresponden a
cada key.
- …: lo último que especificamos son las columnas que vamos a apilar, la notación para seleccionarlas es la misma que usamos con
select().
pew_tidy <- gather(data = pew, income, frequency, -religion)
pew_tidy
#> # A tibble: 90 x 3
#> religion income frequency
#> <chr> <chr> <dbl>
#> 1 Jewish <$10k 19
#> 2 Mainline Prot <$10k 289
#> 3 Mormon <$10k 29
#> 4 Muslim <$10k 6
#> 5 Orthodox <$10k 13
#> 6 Other Christian <$10k 9
#> 7 Other Faiths <$10k 20
#> 8 Other World Religions <$10k 5
#> 9 Unaffiliated <$10k 217
#> 10 Jewish $10-20k 19
#> # ... with 80 more rowsObservemos que en la tabla ancha teníamos bajo la columna <$10k, en el renglón correspondiente a Agnostic un valor de 27, y podemos ver que este valor en la tabla larga se almacena bajo la columna frecuencia y corresponde a religión Agnostic, income <$10k. También es importante ver que en este ejemplo especificamos las columnas a apilar identificando la que no vamos a alargar con un signo negativo: es decir apila todas las columnas menos religión.
La nueva estructura de la base de datos nos permite, por ejemplo, hacer fácilmente una gráfica donde podemos comparar las diferencias en las frecuencias.
Nota: En esta sección no explicaremos las funciones de graficación pues estas se cubren en las notas introductorias a R. En esta parte nos queremos concentrar en como limpiar datos y ejemplificar lo sencillo que es trabajar con datos limpios, esto es, una vez que los datos fueron reestructurados es fácil construir gráficas y resúmenes.
ggplot(pew_tidy, aes(x = income, y = frequency, color = religion, group = religion)) +
geom_line() +
theme(axis.text.x = element_text(angle = 90, hjust = 1))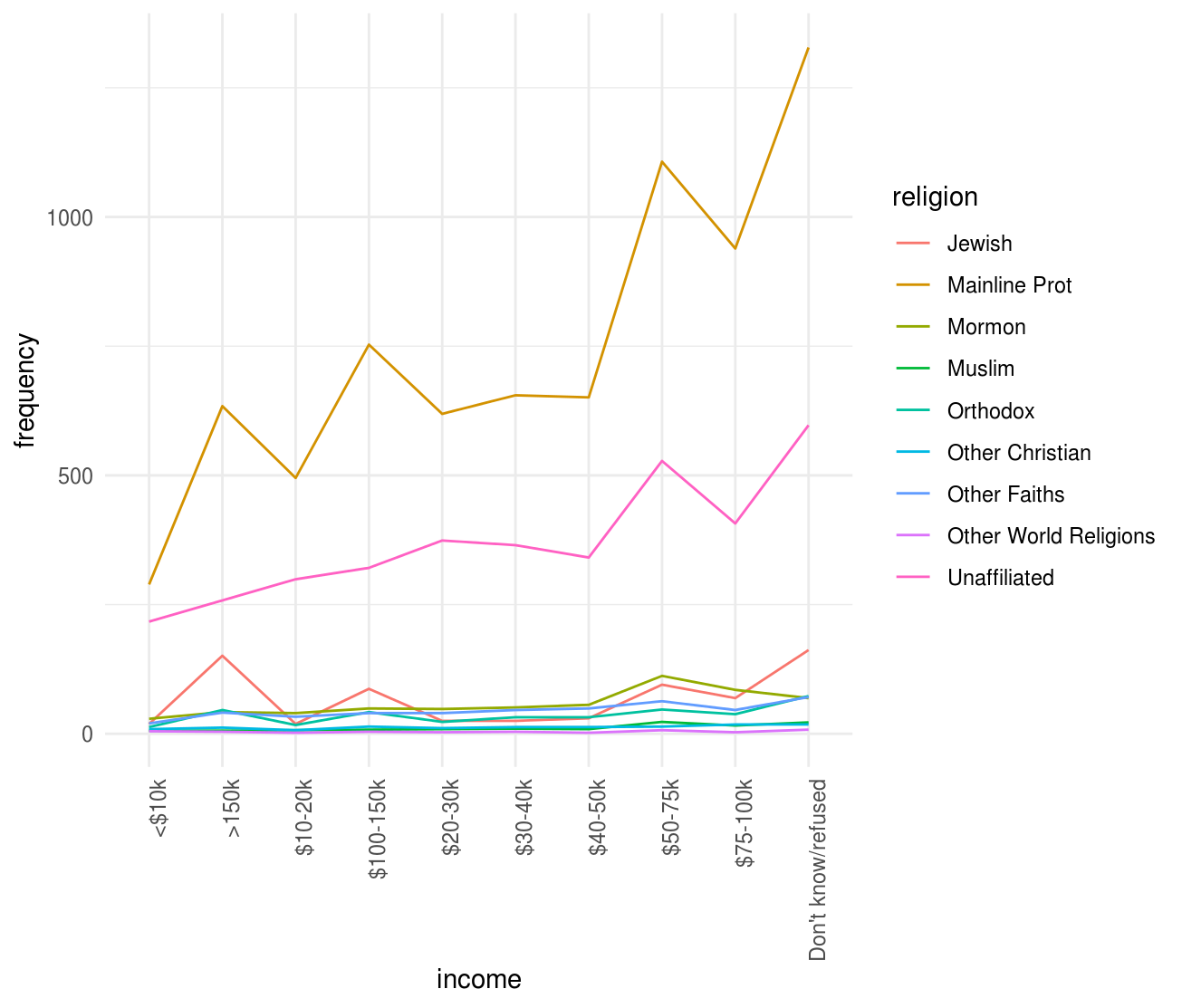
Podemos hacer gráficas más interesantes si creamos nuevas variables:
by_religion <- group_by(pew_tidy, religion)
pew_tidy_2 <- pew_tidy %>%
filter(income != "Don't know/refused") %>%
group_by(religion) %>%
mutate(percent = frequency / sum(frequency)) %>%
filter(sum(frequency) > 1000)
head(pew_tidy_2)
#> # A tibble: 6 x 4
#> # Groups: religion [2]
#> religion income frequency percent
#> <chr> <chr> <dbl> <dbl>
#> 1 Mainline Prot <$10k 289 0.0471
#> 2 Unaffiliated <$10k 217 0.0698
#> 3 Mainline Prot $10-20k 495 0.0806
#> 4 Unaffiliated $10-20k 299 0.0961
#> 5 Mainline Prot $20-30k 619 0.101
#> 6 Unaffiliated $20-30k 374 0.120
income_levels <- unique(pew_tidy$income)[1:9]
ggplot(pew_tidy_2, aes(x = income, y = percent, group = religion)) +
facet_wrap(~ religion, nrow = 1) +
geom_bar(stat = "identity", fill = "darkgray") +
theme(axis.text.x = element_text(angle = 90, hjust = 1)) +
scale_x_discrete(limits = income_levels)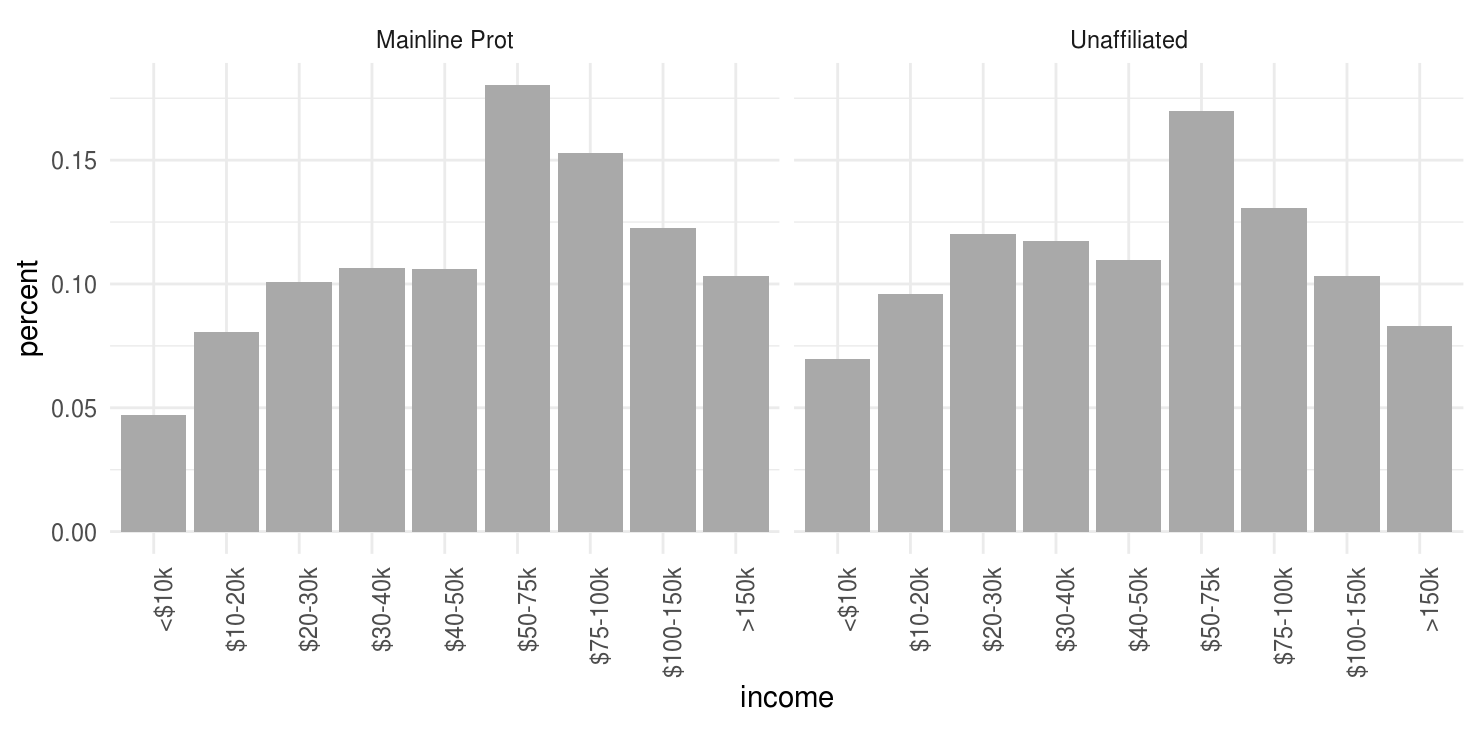
En el código de arriba utilizamos las funciones group_by, filter y mutate
que estudiaremos más adelante. Por ahora concentremonos en gather y spread.
Otro ejemplo, veamos los datos de Billboard, aquí se registra la fecha en la que una canción entra por primera vez al top 100 de Billboard.
billboard <- read_csv("data/billboard.csv")
#> Parsed with column specification:
#> cols(
#> .default = col_double(),
#> artist = col_character(),
#> track = col_character(),
#> time = col_time(format = ""),
#> date.entered = col_date(format = ""),
#> wk66 = col_logical(),
#> wk67 = col_logical(),
#> wk68 = col_logical(),
#> wk69 = col_logical(),
#> wk70 = col_logical(),
#> wk71 = col_logical(),
#> wk72 = col_logical(),
#> wk73 = col_logical(),
#> wk74 = col_logical(),
#> wk75 = col_logical(),
#> wk76 = col_logical()
#> )
#> See spec(...) for full column specifications.
billboard
#> # A tibble: 317 x 81
#> year artist track time date.entered wk1 wk2 wk3 wk4 wk5
#> <dbl> <chr> <chr> <tim> <date> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 2000 2 Pac Baby… 04:22 2000-02-26 87 82 72 77 87
#> 2 2000 2Ge+h… The … 03:15 2000-09-02 91 87 92 NA NA
#> 3 2000 3 Doo… Kryp… 03:53 2000-04-08 81 70 68 67 66
#> 4 2000 3 Doo… Loser 04:24 2000-10-21 76 76 72 69 67
#> 5 2000 504 B… Wobb… 03:35 2000-04-15 57 34 25 17 17
#> 6 2000 98^0 Give… 03:24 2000-08-19 51 39 34 26 26
#> 7 2000 A*Tee… Danc… 03:44 2000-07-08 97 97 96 95 100
#> 8 2000 Aaliy… I Do… 04:15 2000-01-29 84 62 51 41 38
#> 9 2000 Aaliy… Try … 04:03 2000-03-18 59 53 38 28 21
#> 10 2000 Adams… Open… 05:30 2000-08-26 76 76 74 69 68
#> # ... with 307 more rows, and 71 more variables: wk6 <dbl>, wk7 <dbl>,
#> # wk8 <dbl>, wk9 <dbl>, wk10 <dbl>, wk11 <dbl>, wk12 <dbl>, wk13 <dbl>,
#> # wk14 <dbl>, wk15 <dbl>, wk16 <dbl>, wk17 <dbl>, wk18 <dbl>,
#> # wk19 <dbl>, wk20 <dbl>, wk21 <dbl>, wk22 <dbl>, wk23 <dbl>,
#> # wk24 <dbl>, wk25 <dbl>, wk26 <dbl>, wk27 <dbl>, wk28 <dbl>,
#> # wk29 <dbl>, wk30 <dbl>, wk31 <dbl>, wk32 <dbl>, wk33 <dbl>,
#> # wk34 <dbl>, wk35 <dbl>, wk36 <dbl>, wk37 <dbl>, wk38 <dbl>,
#> # wk39 <dbl>, wk40 <dbl>, wk41 <dbl>, wk42 <dbl>, wk43 <dbl>,
#> # wk44 <dbl>, wk45 <dbl>, wk46 <dbl>, wk47 <dbl>, wk48 <dbl>,
#> # wk49 <dbl>, wk50 <dbl>, wk51 <dbl>, wk52 <dbl>, wk53 <dbl>,
#> # wk54 <dbl>, wk55 <dbl>, wk56 <dbl>, wk57 <dbl>, wk58 <dbl>,
#> # wk59 <dbl>, wk60 <dbl>, wk61 <dbl>, wk62 <dbl>, wk63 <dbl>,
#> # wk64 <dbl>, wk65 <dbl>, wk66 <lgl>, wk67 <lgl>, wk68 <lgl>,
#> # wk69 <lgl>, wk70 <lgl>, wk71 <lgl>, wk72 <lgl>, wk73 <lgl>,
#> # wk74 <lgl>, wk75 <lgl>, wk76 <lgl>Notemos que el rank en cada semana (una vez que entró a la lista) está guardado
en 75 columnas wk1 a wk75, este tipo de almacenamiento no es limpio pero
puede ser útil al momento de ingresar la información.
Para tener datos limpios apilamos las semanas de manera que sea una sola columna (nuevamente alargamos los datos):
billboard_long <- gather(billboard, week, rank, wk1:wk76, na.rm = TRUE)
billboard_long
#> # A tibble: 5,307 x 7
#> year artist track time date.entered week rank
#> * <dbl> <chr> <chr> <tim> <date> <chr> <dbl>
#> 1 2000 2 Pac Baby Don't Cry (Kee… 04:22 2000-02-26 wk1 87
#> 2 2000 2Ge+her The Hardest Part Of… 03:15 2000-09-02 wk1 91
#> 3 2000 3 Doors Down Kryptonite 03:53 2000-04-08 wk1 81
#> 4 2000 3 Doors Down Loser 04:24 2000-10-21 wk1 76
#> 5 2000 504 Boyz Wobble Wobble 03:35 2000-04-15 wk1 57
#> 6 2000 98^0 Give Me Just One Ni… 03:24 2000-08-19 wk1 51
#> 7 2000 A*Teens Dancing Queen 03:44 2000-07-08 wk1 97
#> 8 2000 Aaliyah I Don't Wanna 04:15 2000-01-29 wk1 84
#> 9 2000 Aaliyah Try Again 04:03 2000-03-18 wk1 59
#> 10 2000 Adams, Yolan… Open My Heart 05:30 2000-08-26 wk1 76
#> # ... with 5,297 more rowsNotemos que en esta ocasión especificamos las columnas que vamos a apilar
indicando el nombre de la primera de ellas seguido de : y por último el
nombre de la última variable a apilar. Por otra parte, la instrucción
na.rm = TRUE se utiliza para eliminar los renglones con valores faltantes en
la columna de value (rank), esto es, eliminamos aquellas observaciones que
tenían NA en la columnas wknum de la tabla ancha. Ahora realizamos una
limpieza adicional creando mejores variables de fecha.
billboard_tidy <- billboard_long %>%
mutate(
week = parse_number(week),
date = date.entered + 7 * (week - 1),
rank = as.numeric(rank)
) %>%
select(-date.entered)
billboard_tidy
#> # A tibble: 5,307 x 7
#> year artist track time week rank date
#> <dbl> <chr> <chr> <tim> <dbl> <dbl> <date>
#> 1 2000 2 Pac Baby Don't Cry (Keep… 04:22 1 87 2000-02-26
#> 2 2000 2Ge+her The Hardest Part Of … 03:15 1 91 2000-09-02
#> 3 2000 3 Doors Down Kryptonite 03:53 1 81 2000-04-08
#> 4 2000 3 Doors Down Loser 04:24 1 76 2000-10-21
#> 5 2000 504 Boyz Wobble Wobble 03:35 1 57 2000-04-15
#> 6 2000 98^0 Give Me Just One Nig… 03:24 1 51 2000-08-19
#> 7 2000 A*Teens Dancing Queen 03:44 1 97 2000-07-08
#> 8 2000 Aaliyah I Don't Wanna 04:15 1 84 2000-01-29
#> 9 2000 Aaliyah Try Again 04:03 1 59 2000-03-18
#> 10 2000 Adams, Yolanda Open My Heart 05:30 1 76 2000-08-26
#> # ... with 5,297 more rowsNuevamente, podemos hacer gráficas facilmente.
tracks <- filter(billboard_tidy, track %in%
c("Higher", "Amazed", "Kryptonite", "Breathe", "With Arms Wide Open"))
ggplot(tracks, aes(x = date, y = rank)) +
geom_line() +
facet_wrap(~track, nrow = 1) +
theme(axis.text.x = element_text(angle = 90, hjust = 1))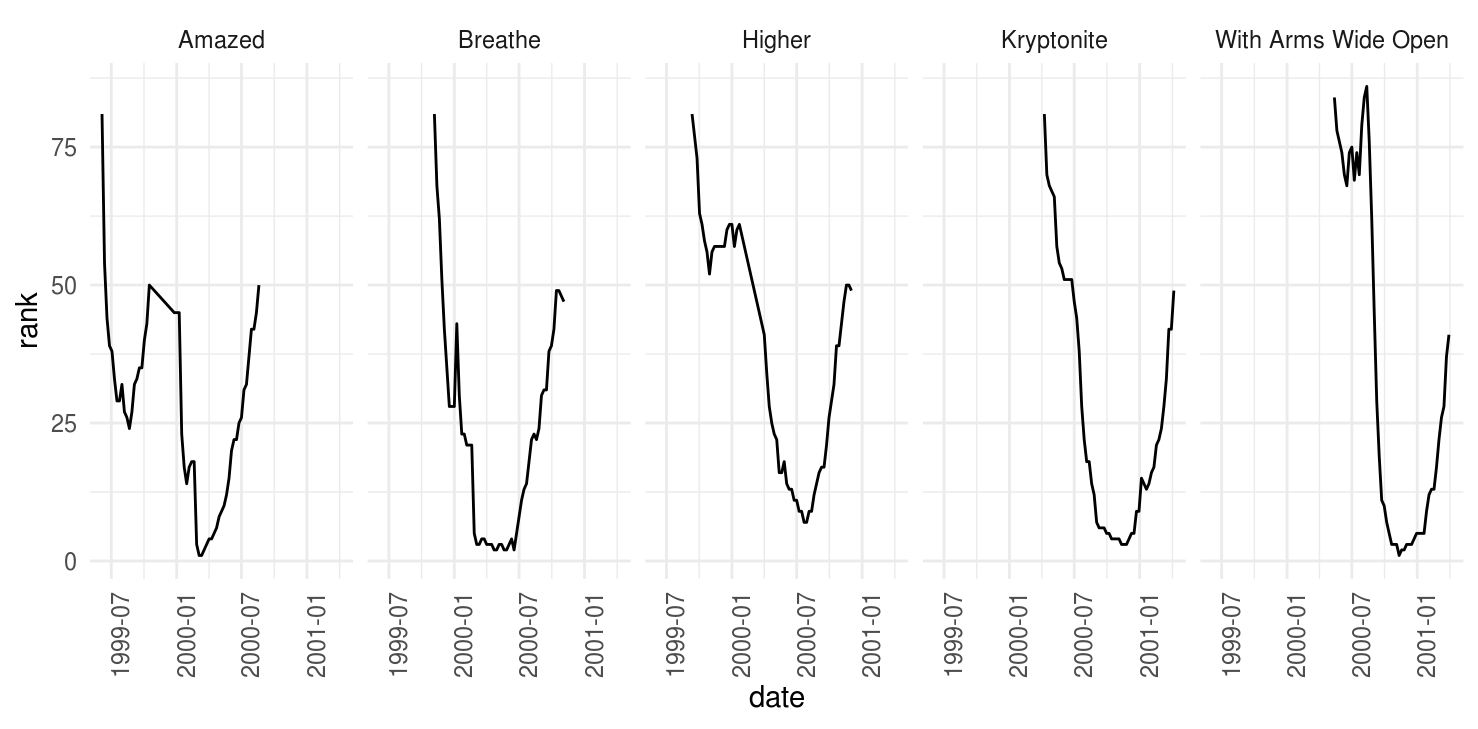
Una columna asociada a más de una variable
La siguiente base de datos proviene de la Organización Mundial de la Salud y contiene el número de casos confirmados de tuberculosis por país y año, la información esta por grupo demográfico de acuerdo a sexo (m, f), y edad (0-4, 5-14, etc). Los datos están disponibles en http://www.who.int/tb/country/data/download/en/.
tb <- read.csv("data/tb.csv") %>% tbl_df()
tb
#> # A tibble: 5,769 x 22
#> iso2 year new_sp_m04 new_sp_m514 new_sp_m014 new_sp_m1524 new_sp_m2534
#> * <fct> <int> <int> <int> <int> <int> <int>
#> 1 AD 1989 NA NA NA NA NA
#> 2 AD 1990 NA NA NA NA NA
#> 3 AD 1991 NA NA NA NA NA
#> 4 AD 1992 NA NA NA NA NA
#> 5 AD 1993 NA NA NA NA NA
#> 6 AD 1994 NA NA NA NA NA
#> 7 AD 1996 NA NA 0 0 0
#> 8 AD 1997 NA NA 0 0 1
#> 9 AD 1998 NA NA 0 0 0
#> 10 AD 1999 NA NA 0 0 0
#> # ... with 5,759 more rows, and 15 more variables: new_sp_m3544 <int>,
#> # new_sp_m4554 <int>, new_sp_m5564 <int>, new_sp_m65 <int>,
#> # new_sp_mu <int>, new_sp_f04 <int>, new_sp_f514 <int>,
#> # new_sp_f014 <int>, new_sp_f1524 <int>, new_sp_f2534 <int>,
#> # new_sp_f3544 <int>, new_sp_f4554 <int>, new_sp_f5564 <int>,
#> # new_sp_f65 <int>, new_sp_fu <int> De manera similar a los ejemplos anteriores,
utiliza la función
De manera similar a los ejemplos anteriores,
utiliza la función gather para apilar las columnas correspondientes a
sexo-edad.
Piensa en como podemos separar la “variable” sexo-edad en dos columnas.
Ahora separaremos las variables sexo y edad de la columna demo, para ello
debemos pasar a la función separate(), esta recibe como parámetros:
el nombre de la base de datos,
el nombre de la variable que deseamos separar en más de una,
la posición de donde deseamos “cortar” (hay más opciones para especificar como separar, ver
?separate). El default es separar valores en todos los lugares que encuentre un caracter que no es alfanumérico (espacio, guión,…).
tb_tidy <- separate(tb_long, demo, c("sex", "age"), 8)
tb_tidy
#> # A tibble: 35,750 x 5
#> iso2 year sex age n
#> * <fct> <int> <chr> <chr> <int>
#> 1 AD 2005 new_sp_m 04 0
#> 2 AD 2006 new_sp_m 04 0
#> 3 AD 2008 new_sp_m 04 0
#> 4 AE 2006 new_sp_m 04 0
#> 5 AE 2007 new_sp_m 04 0
#> 6 AE 2008 new_sp_m 04 0
#> 7 AG 2007 new_sp_m 04 0
#> 8 AL 2005 new_sp_m 04 0
#> 9 AL 2006 new_sp_m 04 1
#> 10 AL 2007 new_sp_m 04 0
#> # ... with 35,740 more rows
table(tb_tidy$sex)
#>
#> new_sp_f new_sp_m
#> 17830 17920
# creamos un mejor código de genero
tb_tidy <- mutate(tb_tidy, sex = substr(sex, 8, 8))
table(tb_tidy$sex)
#>
#> f m
#> 17830 17920Variables almacenadas en filas y columnas
El problema más difícil es cuando las variables están tanto en filas como en columnas, veamos una base de datos de clima en Cuernavaca. ¿Cuáles son las variables en estos datos?
clima <- read_delim("data/clima.txt", "\t", escape_double = FALSE,
trim_ws = TRUE)
#> Parsed with column specification:
#> cols(
#> .default = col_double(),
#> id = col_character(),
#> element = col_character(),
#> d9 = col_logical(),
#> d12 = col_logical(),
#> d18 = col_logical(),
#> d19 = col_logical(),
#> d20 = col_logical(),
#> d21 = col_logical(),
#> d22 = col_logical(),
#> d24 = col_logical()
#> )
#> See spec(...) for full column specifications.Estos datos tienen variables en columnas individuales (id, año, mes), en múltiples columnas (día, d1-d31) y en filas (tmin, tmax). Comencemos por apilar las columnas.
clima_long <- gather(clima, day, value, d1:d31, na.rm = TRUE)
clima_long
#> # A tibble: 66 x 6
#> id year month element day value
#> * <chr> <dbl> <dbl> <chr> <chr> <dbl>
#> 1 MX000017004 2010 12 TMAX d1 299
#> 2 MX000017004 2010 12 TMIN d1 138
#> 3 MX000017004 2010 2 TMAX d2 273
#> 4 MX000017004 2010 2 TMIN d2 144
#> 5 MX000017004 2010 11 TMAX d2 313
#> 6 MX000017004 2010 11 TMIN d2 163
#> 7 MX000017004 2010 2 TMAX d3 241
#> 8 MX000017004 2010 2 TMIN d3 144
#> 9 MX000017004 2010 7 TMAX d3 286
#> 10 MX000017004 2010 7 TMIN d3 175
#> # ... with 56 more rowsPodemos crear algunas variables adicionales.
clima_vars <- clima_long %>%
mutate(day = parse_number(day),
value = as.numeric(value) / 10) %>%
select(id, year, month, day, element, value) %>%
arrange(id, year, month, day)
clima_vars
#> # A tibble: 66 x 6
#> id year month day element value
#> <chr> <dbl> <dbl> <dbl> <chr> <dbl>
#> 1 MX000017004 2010 1 30 TMAX 27.8
#> 2 MX000017004 2010 1 30 TMIN 14.5
#> 3 MX000017004 2010 2 2 TMAX 27.3
#> 4 MX000017004 2010 2 2 TMIN 14.4
#> 5 MX000017004 2010 2 3 TMAX 24.1
#> 6 MX000017004 2010 2 3 TMIN 14.4
#> 7 MX000017004 2010 2 11 TMAX 29.7
#> 8 MX000017004 2010 2 11 TMIN 13.4
#> 9 MX000017004 2010 2 23 TMAX 29.9
#> 10 MX000017004 2010 2 23 TMIN 10.7
#> # ... with 56 more rowsFinalmente, la columna element no es una variable, sino que almacena el nombre
de dos variables, la operación que debemos aplicar (spread) es el inverso de
apilar (gather):
clima_tidy <- spread(clima_vars, element, value)
clima_tidy
#> # A tibble: 33 x 6
#> id year month day TMAX TMIN
#> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
#> 1 MX000017004 2010 1 30 27.8 14.5
#> 2 MX000017004 2010 2 2 27.3 14.4
#> 3 MX000017004 2010 2 3 24.1 14.4
#> 4 MX000017004 2010 2 11 29.7 13.4
#> 5 MX000017004 2010 2 23 29.9 10.7
#> 6 MX000017004 2010 3 5 32.1 14.2
#> 7 MX000017004 2010 3 10 34.5 16.8
#> 8 MX000017004 2010 3 16 31.1 17.6
#> 9 MX000017004 2010 4 27 36.3 16.7
#> 10 MX000017004 2010 5 27 33.2 18.2
#> # ... with 23 more rowsAhora es inmediato no solo hacer gráficas sino también ajustar un modelo.
# ajustamos un modelo lineal donde la variable respuesta es temperatura
# máxima, y la variable explicativa es el mes
clima_lm <- lm(TMAX ~ factor(month), data = clima_tidy)
summary(clima_lm)
#>
#> Call:
#> lm(formula = TMAX ~ factor(month), data = clima_tidy)
#>
#> Residuals:
#> Min 1Q Median 3Q Max
#> -3.65 -0.92 -0.02 1.05 3.18
#>
#> Coefficients:
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) 27.8000 1.8610 14.938 5.34e-13 ***
#> factor(month)2 -0.0500 2.0807 -0.024 0.98104
#> factor(month)3 4.7667 2.1489 2.218 0.03717 *
#> factor(month)4 8.5000 2.6319 3.230 0.00385 **
#> factor(month)5 5.4000 2.6319 2.052 0.05228 .
#> factor(month)6 1.2500 2.2793 0.548 0.58892
#> factor(month)7 1.4500 2.2793 0.636 0.53123
#> factor(month)8 0.4714 1.9895 0.237 0.81488
#> factor(month)10 1.1000 2.0386 0.540 0.59491
#> factor(month)11 0.3200 2.0386 0.157 0.87670
#> factor(month)12 1.0500 2.2793 0.461 0.64955
#> ---
#> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#>
#> Residual standard error: 1.861 on 22 degrees of freedom
#> Multiple R-squared: 0.6182, Adjusted R-squared: 0.4447
#> F-statistic: 3.563 on 10 and 22 DF, p-value: 0.006196Mas de un tipo de observación en una misma tabla
En ocasiones las bases de datos involucran valores en diferentes niveles, en diferentes tipos de unidad observacional. En la limpieza de datos, cada unidad observacional debe estar almacenada en su propia tabla (esto esta ligado a normalización de una base de datos), es importante para evitar inconsistencias en los datos.
¿Cuáles son las unidades observacionales de los datos de billboard?
billboard_tidy
#> # A tibble: 5,307 x 7
#> year artist track time week rank date
#> <dbl> <chr> <chr> <tim> <dbl> <dbl> <date>
#> 1 2000 2 Pac Baby Don't Cry (Keep… 04:22 1 87 2000-02-26
#> 2 2000 2Ge+her The Hardest Part Of … 03:15 1 91 2000-09-02
#> 3 2000 3 Doors Down Kryptonite 03:53 1 81 2000-04-08
#> 4 2000 3 Doors Down Loser 04:24 1 76 2000-10-21
#> 5 2000 504 Boyz Wobble Wobble 03:35 1 57 2000-04-15
#> 6 2000 98^0 Give Me Just One Nig… 03:24 1 51 2000-08-19
#> 7 2000 A*Teens Dancing Queen 03:44 1 97 2000-07-08
#> 8 2000 Aaliyah I Don't Wanna 04:15 1 84 2000-01-29
#> 9 2000 Aaliyah Try Again 04:03 1 59 2000-03-18
#> 10 2000 Adams, Yolanda Open My Heart 05:30 1 76 2000-08-26
#> # ... with 5,297 more rowsSeparemos esta base de datos en dos: la tabla canción que almacena artista, nombre de la canción y duración; la tabla rank que almacena el ranking de la canción en cada semana.
song <- billboard_tidy %>%
select(artist, track, year, time) %>%
unique() %>%
arrange(artist) %>%
mutate(song_id = row_number(artist))
song
#> # A tibble: 317 x 5
#> artist track year time song_id
#> <chr> <chr> <dbl> <time> <int>
#> 1 2 Pac Baby Don't Cry (Keep... 2000 04:22 1
#> 2 2Ge+her The Hardest Part Of ... 2000 03:15 2
#> 3 3 Doors Down Kryptonite 2000 03:53 3
#> 4 3 Doors Down Loser 2000 04:24 4
#> 5 504 Boyz Wobble Wobble 2000 03:35 5
#> 6 98^0 Give Me Just One Nig... 2000 03:24 6
#> 7 A*Teens Dancing Queen 2000 03:44 7
#> 8 Aaliyah I Don't Wanna 2000 04:15 8
#> 9 Aaliyah Try Again 2000 04:03 9
#> 10 Adams, Yolanda Open My Heart 2000 05:30 10
#> # ... with 307 more rows
rank <- billboard_tidy %>%
left_join(song, c("artist", "track", "year", "time")) %>%
select(song_id, date, week, rank) %>%
arrange(song_id, date) %>%
tbl_df
rank
#> # A tibble: 5,307 x 4
#> song_id date week rank
#> <int> <date> <dbl> <dbl>
#> 1 1 2000-02-26 1 87
#> 2 1 2000-03-04 2 82
#> 3 1 2000-03-11 3 72
#> 4 1 2000-03-18 4 77
#> 5 1 2000-03-25 5 87
#> 6 1 2000-04-01 6 94
#> 7 1 2000-04-08 7 99
#> 8 2 2000-09-02 1 91
#> 9 2 2000-09-09 2 87
#> 10 2 2000-09-16 3 92
#> # ... with 5,297 more rowsUna misma unidad observacional está almacenada en múltiples tablas
También es común que los valores sobre una misma unidad observacional estén separados en muchas tablas o archivos, es común que estas tablas esten divididas de acuerdo a una variable, de tal manera que cada archivo representa a una persona, año o ubicación. Para juntar los archivos hacemos lo siguiente:
- Leemos los archivos en una lista de tablas.
- Para cada tabla agregamos una columna que registra el nombre del archivo original.
- Combinamos las tablas en un solo data frame.
Veamos un ejemplo, descarga la carpeta specdata, ésta contiene 332 archivos csv que almacenan información de monitoreo de contaminación en 332 ubicaciones de EUA. Cada archivo contiene información de una unidad de monitoreo y el número de identificación del monitor es el nombre del archivo.
Los pasos en R (usando el paquete purrr), primero creamos un vector con los
nombres de los archivos en un directorio, eligiendo aquellos que contengan las
letras “.csv”.
paths <- dir("data/specdata", pattern = "\\.csv$", full.names = TRUE) Después le asignamos el nombre del csv al nombre de cada elemento del vector. Este paso se realiza para preservar los nombres de los archivos ya que estos los asignaremos a una variable mas adelante.
paths <- set_names(paths, basename(paths))La función map_df itera sobre cada dirección, lee el csv en dicha dirección y
los combina en un data frame.
specdata_us <- map_df(paths, ~read_csv(., col_types = "Tddi"), .id = "filename")
# eliminamos la basura del id
specdata <- specdata_us %>%
mutate(monitor = parse_number(filename)) %>%
select(id = ID, monitor, date = Date, sulfate, nitrate)
specdata
#> # A tibble: 772,087 x 5
#> id monitor date sulfate nitrate
#> <int> <dbl> <dttm> <dbl> <dbl>
#> 1 1 1 2003-01-01 00:00:00 NA NA
#> 2 1 1 2003-01-02 00:00:00 NA NA
#> 3 1 1 2003-01-03 00:00:00 NA NA
#> 4 1 1 2003-01-04 00:00:00 NA NA
#> 5 1 1 2003-01-05 00:00:00 NA NA
#> 6 1 1 2003-01-06 00:00:00 NA NA
#> 7 1 1 2003-01-07 00:00:00 NA NA
#> 8 1 1 2003-01-08 00:00:00 NA NA
#> 9 1 1 2003-01-09 00:00:00 NA NA
#> 10 1 1 2003-01-10 00:00:00 NA NA
#> # ... with 772,077 more rowsOtras consideraciones
En las buenas prácticas es importante tomar en cuenta los siguientes puntos:
Incluir un encabezado con el nombre de las variables.
Los nombres de las variables deben ser entendibles (e.g. AgeAtDiagnosis es mejor que AgeDx).
En general los datos se deben guardar en un archivo por tabla.
Escribir un script con las modificaciones que se hicieron a los datos crudos (reproducibilidad).
Otros aspectos importantes en la limpieza de datos son: selección del tipo de variables (por ejemplo fechas), datos faltantes, typos y detección de valores atípicos.
Recursos adicionales
Data Import Cheat Sheet, RStudio.
Data Transformation Cheat Sheet, RStudio.
Referencias
Wickham, Hadley. 2014. “Tidy Data.” Journal of Statistical Software, Articles 59 (10):1–23. https://doi.org/10.18637/jss.v059.i10.