3.1 Transformación de datos
Separa-aplica-combina (split-apply-combine)
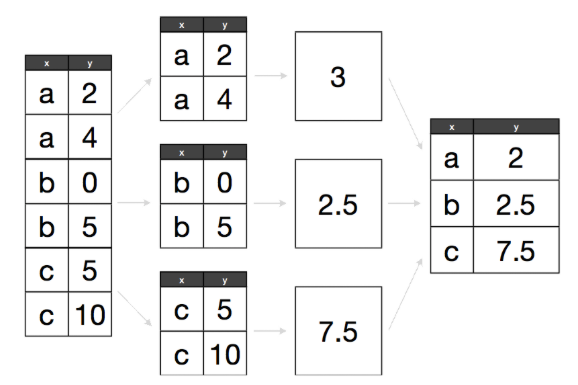
Muchos problemas de análisis de datos involucran la aplicación de la estrategia separa-aplica-combina (Wickham 2011), esta consiste en romper un problema en pedazos (de acuerdo a una variable de interés), operar sobre cada subconjunto de manera independiente (ej. calcular la media de cada grupo, ordenar observaciones por grupo, estandarizar por grupo) y después unir los pedazos nuevamente. El siguiente diagrama ejemplifiaca el paradigma de divide-aplica-combina:
- Separa la base de datos original.
- Aplica funciones a cada subconjunto.
- Combina los resultados en una nueva base de datos.
Ahora, cuando pensamos como implementar la estrategia divide-aplica-combina es
natural pensar en iteraciones, por ejemplo utilizar un ciclo for para recorrer
cada grupo de interés y aplicar las funciones, sin embargo la aplicación de
ciclos for desemboca en código difícil de entender por lo que preferimos
trabajar con funciones creadas para estas tareas, usaremos el paquete
dplyr que además de ser más claro suele ser más veloz.
Estudiaremos las siguientes funciones:
- filter: obten un subconjunto de las filas de acuerdo a un criterio.
- select: selecciona columnas de acuerdo al nombre
- arrange: reordena las filas
- mutate: agrega nuevas variables
- summarise: reduce variables a valores (crear nuevas bases de datos con resúmenes de variables de la base original)
Estas funciones trabajan de manera similar, el primer argumento que reciben es un data frame, los argumentos que siguen indican que operación se va a efectuar y el resultado es un nuevo data frame.
Adicionalmente, se pueden usar con group_by que cambia el dominio de cada función, pasando de operar en el conjunto de datos completos a operar en grupos, esto lo veremos más adelante.
Ejemplos y lectura de datos
En esta sección trabajaremos con bases de datos de vuelos del aeropuerto de Houston. Comenzamos importando los datos a R.
Para leer los datos usamos funciones del paquete readr que forma parte del
tidyverse, notemos que si estamos usando RStudio podemos generar los comandos
de lectura de datos usando la opción Import Dataset en la ventana de
Environment.
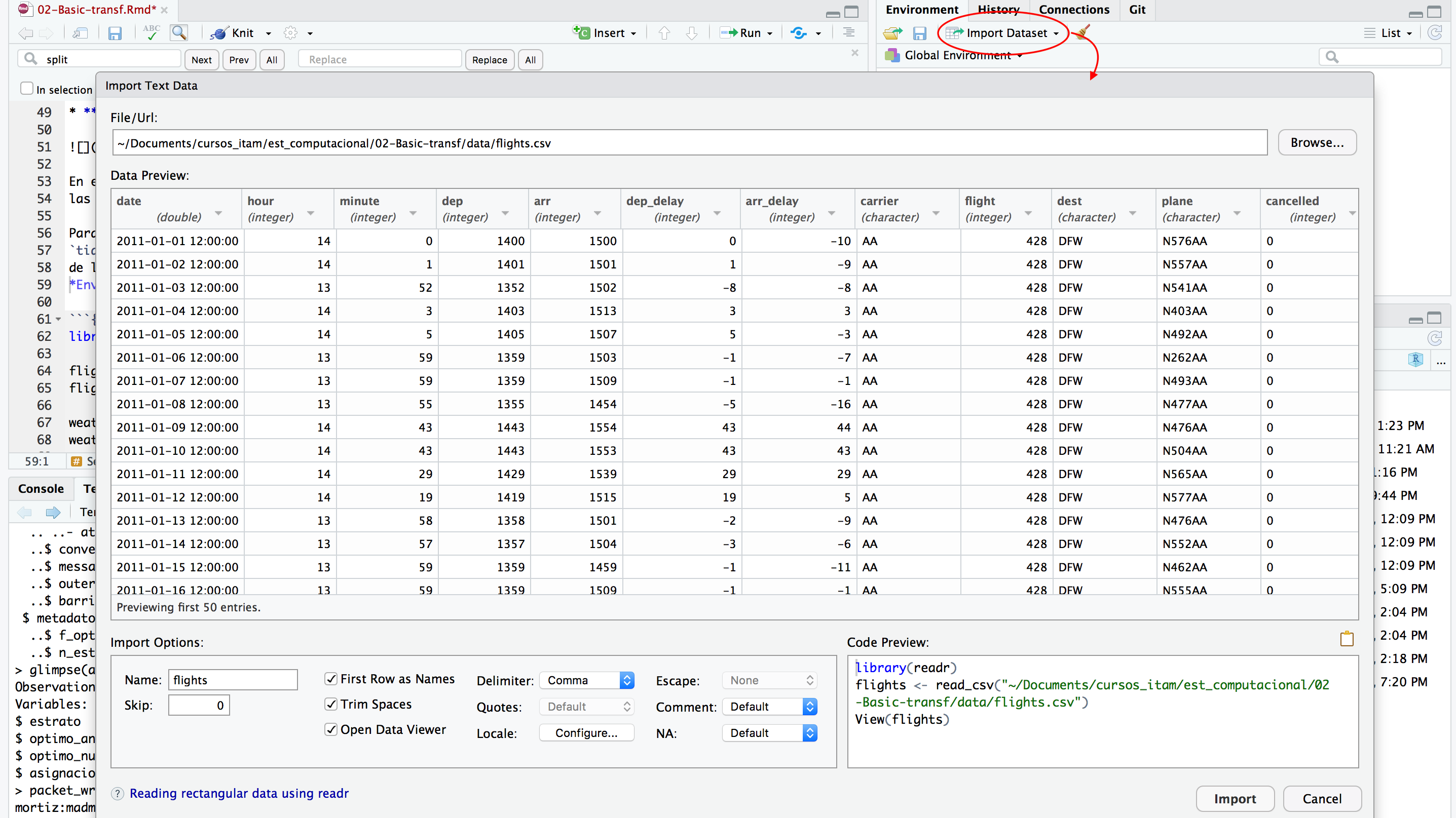
Si usamos la opción de importar datos usando la funcionalidad point-and-click de RStudio, es importante copiar los comandos al script de R para no perder reproducibilidad.
library(tidyverse)
flights <- read_csv("data/flights.csv")
#> Parsed with column specification:
#> cols(
#> date = col_datetime(format = ""),
#> hour = col_double(),
#> minute = col_double(),
#> dep = col_double(),
#> arr = col_double(),
#> dep_delay = col_double(),
#> arr_delay = col_double(),
#> carrier = col_character(),
#> flight = col_double(),
#> dest = col_character(),
#> plane = col_character(),
#> cancelled = col_double(),
#> time = col_double(),
#> dist = col_double()
#> )
flights
#> # A tibble: 227,496 x 14
#> date hour minute dep arr dep_delay arr_delay carrier
#> <dttm> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
#> 1 2011-01-01 12:00:00 14 0 1400 1500 0 -10 AA
#> 2 2011-01-02 12:00:00 14 1 1401 1501 1 -9 AA
#> 3 2011-01-03 12:00:00 13 52 1352 1502 -8 -8 AA
#> 4 2011-01-04 12:00:00 14 3 1403 1513 3 3 AA
#> 5 2011-01-05 12:00:00 14 5 1405 1507 5 -3 AA
#> 6 2011-01-06 12:00:00 13 59 1359 1503 -1 -7 AA
#> 7 2011-01-07 12:00:00 13 59 1359 1509 -1 -1 AA
#> 8 2011-01-08 12:00:00 13 55 1355 1454 -5 -16 AA
#> 9 2011-01-09 12:00:00 14 43 1443 1554 43 44 AA
#> 10 2011-01-10 12:00:00 14 43 1443 1553 43 43 AA
#> # ... with 227,486 more rows, and 6 more variables: flight <dbl>,
#> # dest <chr>, plane <chr>, cancelled <dbl>, time <dbl>, dist <dbl>
weather <- read_csv("data/weather.csv")
#> Parsed with column specification:
#> cols(
#> date = col_date(format = ""),
#> hour = col_double(),
#> temp = col_double(),
#> dew_point = col_double(),
#> humidity = col_double(),
#> pressure = col_double(),
#> visibility = col_double(),
#> wind_dir = col_character(),
#> wind_dir2 = col_double(),
#> wind_speed = col_double(),
#> gust_speed = col_double(),
#> precip = col_double(),
#> conditions = col_character(),
#> events = col_character()
#> )
weather
#> # A tibble: 8,723 x 14
#> date hour temp dew_point humidity pressure visibility wind_dir
#> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
#> 1 2011-01-01 0 59 28.9 32 29.9 10 NNE
#> 2 2011-01-01 1 57.2 28.4 33 29.9 10 NNE
#> 3 2011-01-01 2 55.4 28.4 36 29.9 10 NNW
#> 4 2011-01-01 3 53.6 28.4 38 29.9 10 North
#> 5 2011-01-01 4 NA NA NA 30.0 10 NNW
#> 6 2011-01-01 5 NA NA NA 30.0 10 North
#> 7 2011-01-01 6 53.1 17.1 24 30.0 10 North
#> 8 2011-01-01 7 53.1 16 23 30.1 10 North
#> 9 2011-01-01 8 54 18 24 30.1 10 North
#> 10 2011-01-01 9 55.4 17.6 23 30.1 10 NNE
#> # ... with 8,713 more rows, and 6 more variables: wind_dir2 <dbl>,
#> # wind_speed <dbl>, gust_speed <dbl>, precip <dbl>, conditions <chr>,
#> # events <chr>
planes <- read_csv("data/planes.csv")
#> Parsed with column specification:
#> cols(
#> plane = col_character(),
#> year = col_double(),
#> mfr = col_character(),
#> model = col_character(),
#> no.eng = col_double(),
#> no.seats = col_double(),
#> speed = col_double(),
#> engine = col_character(),
#> type = col_character()
#> )
planes
#> # A tibble: 2,853 x 9
#> plane year mfr model no.eng no.seats speed engine type
#> <chr> <dbl> <chr> <chr> <dbl> <dbl> <dbl> <chr> <chr>
#> 1 N576AA 1991 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 2 N557AA 1993 MARZ BAR… KITFOX… 1 2 NA Recipr… Fixed win…
#> 3 N403AA 1974 RAVEN S55A NA 1 60 None Balloon
#> 4 N492AA 1989 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 5 N262AA 1985 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 6 N493AA 1989 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 7 N477AA 1988 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 8 N476AA 1988 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> 9 N504AA NA AUTHIER … TIERRA… 1 2 NA Recipr… Fixed win…
#> 10 N565AA 1987 MCDONNEL… DC-9-8… 2 172 NA Turbo-… Fixed win…
#> # ... with 2,843 more rows
airports <- read_csv("data/airports.csv")
#> Parsed with column specification:
#> cols(
#> iata = col_character(),
#> airport = col_character(),
#> city = col_character(),
#> state = col_character(),
#> country = col_character(),
#> lat = col_double(),
#> long = col_double()
#> )
airports
#> # A tibble: 3,376 x 7
#> iata airport city state country lat long
#> <chr> <chr> <chr> <chr> <chr> <dbl> <dbl>
#> 1 00M Thigpen Bay Springs MS USA 32.0 -89.2
#> 2 00R Livingston Municipal Livingston TX USA 30.7 -95.0
#> 3 00V Meadow Lake Colorado Springs CO USA 38.9 -105.
#> 4 01G Perry-Warsaw Perry NY USA 42.7 -78.1
#> 5 01J Hilliard Airpark Hilliard FL USA 30.7 -81.9
#> 6 01M Tishomingo County Belmont MS USA 34.5 -88.2
#> 7 02A Gragg-Wade Clanton AL USA 32.9 -86.6
#> 8 02C Capitol Brookfield WI USA 43.1 -88.2
#> 9 02G Columbiana County East Liverpool OH USA 40.7 -80.6
#> 10 03D Memphis Memorial Memphis MO USA 40.4 -92.2
#> # ... with 3,366 more rowsFiltrar
Creamos una base de datos de juguete para mostrar el funcionamiento de cada instrucción:
df_ej <- tibble(genero = c("mujer", "hombre", "mujer", "mujer", "hombre"),
estatura = c(1.65, 1.80, 1.70, 1.60, 1.67))
df_ej
#> # A tibble: 5 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 mujer 1.65
#> 2 hombre 1.8
#> 3 mujer 1.7
#> 4 mujer 1.6
#> 5 hombre 1.67El primer argumento de filter() es el nombre del data frame, los subsecuentes
son las expresiones que indican que filas filtrar.
filter(df_ej, genero == "mujer")
#> # A tibble: 3 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 mujer 1.65
#> 2 mujer 1.7
#> 3 mujer 1.6
filter(df_ej, estatura > 1.65 & estatura < 1.75)
#> # A tibble: 2 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 mujer 1.7
#> 2 hombre 1.67Algunos operadores importantes para filtrar son:
x > 1
x >= 1
x < 1
x <= 1
x != 1
x == 1
x %in% c("a", "b")Debemos tener cuidado al usar ==
sqrt(2) ^ 2 == 2
#> [1] FALSE
1/49 * 49 == 1
#> [1] FALSELos resultados de arriba se deben a que las computadoras usan aritmética de precisión finita:
print(1/49 * 49, digits = 20)
#> [1] 0.99999999999999988898Para estos casos es útil usar la función near()
near(sqrt(2) ^ 2, 2)
#> [1] TRUE
near(1 / 49 * 49, 1)
#> [1] TRUELos operadores booleanos también son convenientes para filtrar:
# Conjuntos
a | b
a & b
a & !b
xor(a, b)El siguiente esquema nos ayuda a entender que hace cada operación:
knitr::include_graphics("imagenes/transform-logical.png")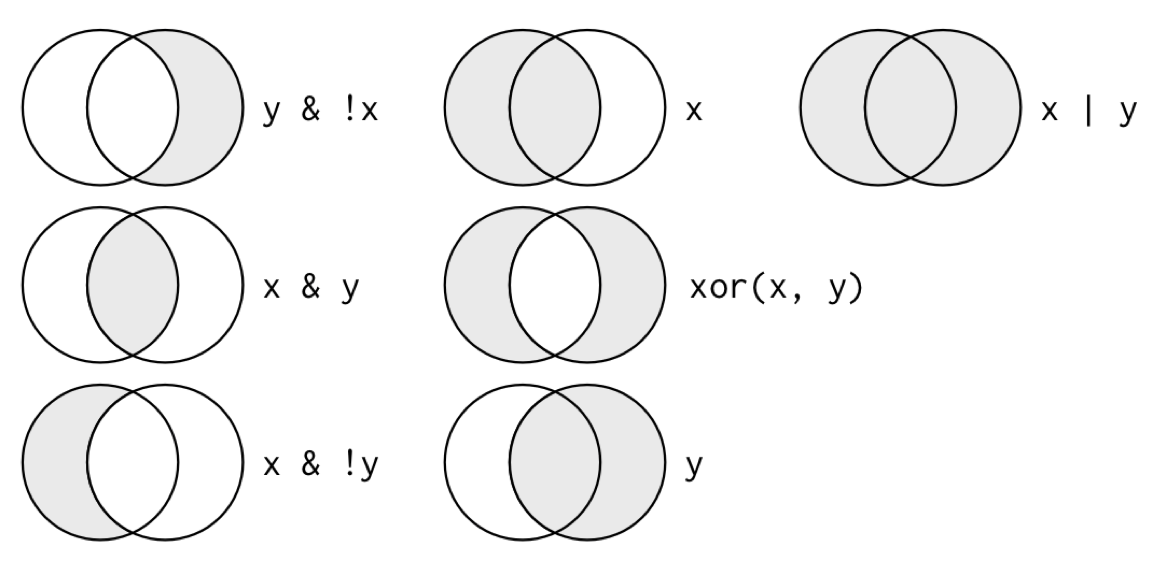
 Encuentra todos los vuelos hacia SFO ó OAK.
Encuentra todos los vuelos hacia SFO ó OAK.
Los vuelos con un retraso mayor a una hora.
En los que el retraso de llegada es más del doble que el retraso de salida.
Un caso común es cuando se desea eliminar los datos con faltantes en una o más
columnas de las tablas de datos, en R los datos faltantes se expresan como NA,
para eliminar los faltantes en la variable dep_delay resulta natural escribir:
filter(flights, dep_delay != NA)
#> # A tibble: 0 x 14
#> # ... with 14 variables: date <dttm>, hour <dbl>, minute <dbl>, dep <dbl>,
#> # arr <dbl>, dep_delay <dbl>, arr_delay <dbl>, carrier <chr>,
#> # flight <dbl>, dest <chr>, plane <chr>, cancelled <dbl>, time <dbl>,
#> # dist <dbl>que nos devuelve una tabla vacía, sin embargo, si hay faltantes en esta
variable. El problema resulta de usar el operador !=, pensemos ¿qué regresan
las siguientes expresiones?
5 + NA
NA / 2
sum(c(5, 4, NA))
mean(c(5, 4, NA))
NA < 3
NA == 3
NA == NALas expresiones anteriores regresan NA, el hecho que la media de un vector
que incluye NAs o su suma regrese NAs se debe a que el default en R es
propagar los valores faltantes, esto es, si deconozco el valor de una de las
componentes de un vector, también desconozco la suma del mismo; sin embargo,
muchas funciones tienen un argumento na.rm para removerlos,
sum(c(5, 4, NA), na.rm = TRUE)
#> [1] 9
mean(c(5, 4, NA), na.rm = TRUE)
#> [1] 4.5Aún queda pendiente, como filtrarlos en una tabla, para esto veamos que el manejo de datos faltantes en R utiliza una lógica ternaria (como SQL):
NA == NA
#> [1] NALa expresión anterior puede resultar confusa, una manera de pensar en esto es considerar los NA como no sé, por ejemplo si no se la edad de Juan y no se la edad de Esteban, la respuesta a ¿Juan tiene la misma edad que Esteban? es no sé (NA).
edad_Juan <- NA
edad_Esteban <- NA
edad_Juan == edad_Esteban
#> [1] NA
edad_Jose <- 32
# Juan es menor que José?
edad_Juan < edad_Jose
#> [1] NAPor tanto para determinar si un valor es faltante usamos la instrucción
is.na().
is.na(NA)
#> [1] TRUEY finalmente podemos filtrar con
filter(flights, is.na(dep_delay))Seleccionar
Elegir columnas de un conjunto de datos.
df_ej
#> # A tibble: 5 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 mujer 1.65
#> 2 hombre 1.8
#> 3 mujer 1.7
#> 4 mujer 1.6
#> 5 hombre 1.67
select(df_ej, genero)
#> # A tibble: 5 x 1
#> genero
#> <chr>
#> 1 mujer
#> 2 hombre
#> 3 mujer
#> 4 mujer
#> 5 hombre
select(df_ej, -genero)
#> # A tibble: 5 x 1
#> estatura
#> <dbl>
#> 1 1.65
#> 2 1.8
#> 3 1.7
#> 4 1.6
#> 5 1.67select(df_ej, starts_with("g"))
select(df_ej, contains("g")) Ve la ayuda de select (
Ve la ayuda de select (?select) y escribe tres
maneras de seleccionar las variables de retraso (delay).
Ordenar
Ordenar de acuerdo al valor de una o más variables:
arrange(df_ej, genero)
#> # A tibble: 5 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 hombre 1.8
#> 2 hombre 1.67
#> 3 mujer 1.65
#> 4 mujer 1.7
#> 5 mujer 1.6
arrange(df_ej, desc(estatura))
#> # A tibble: 5 x 2
#> genero estatura
#> <chr> <dbl>
#> 1 hombre 1.8
#> 2 mujer 1.7
#> 3 hombre 1.67
#> 4 mujer 1.65
#> 5 mujer 1.6 Ordena los vuelos por fecha de salida y hora.
Ordena los vuelos por fecha de salida y hora.
¿Cuáles son los vuelos con mayor retraso?
¿Qué vuelos ganaron más tiempo en el aire?
Mutar
Mutar consiste en crear nuevas variables aplicando una función a columnas existentes:
mutate(df_ej, estatura_cm = estatura * 100)
#> # A tibble: 5 x 3
#> genero estatura estatura_cm
#> <chr> <dbl> <dbl>
#> 1 mujer 1.65 165
#> 2 hombre 1.8 180
#> 3 mujer 1.7 170
#> 4 mujer 1.6 160
#> 5 hombre 1.67 167
mutate(df_ej, estatura_cm = estatura * 100, estatura_in = estatura_cm * 0.3937)
#> # A tibble: 5 x 4
#> genero estatura estatura_cm estatura_in
#> <chr> <dbl> <dbl> <dbl>
#> 1 mujer 1.65 165 65.0
#> 2 hombre 1.8 180 70.9
#> 3 mujer 1.7 170 66.9
#> 4 mujer 1.6 160 63.0
#> 5 hombre 1.67 167 65.7 Calcula la velocidad en millas por hora a partir de
la variable tiempo y la distancia (en millas). ¿Quá vuelo fue el más rápido?
Calcula la velocidad en millas por hora a partir de
la variable tiempo y la distancia (en millas). ¿Quá vuelo fue el más rápido?
Crea una nueva variable que muestre cuánto tiempo se ganó o perdió durante el vuelo.
Hay muchas funciones que podemos usar para crear nuevas variables con mutate(), éstas deben cumplir ser funciones vectorizadas, es decir, reciben un vector de valores y devuelven un vector de la misma dimensión.
Summarise y resúmenes por grupo
Summarise sirve para crear nuevas bases de datos con resúmenes o agregaciones de los datos originales.
summarise(df_ej, promedio = mean(estatura))
#> # A tibble: 1 x 1
#> promedio
#> <dbl>
#> 1 1.68Podemos hacer resúmenes por grupo, primero creamos una base de datos agrupada:
by_genero <- group_by(df_ej, genero)
by_genero
#> # A tibble: 5 x 2
#> # Groups: genero [2]
#> genero estatura
#> <chr> <dbl>
#> 1 mujer 1.65
#> 2 hombre 1.8
#> 3 mujer 1.7
#> 4 mujer 1.6
#> 5 hombre 1.67y después operamos sobre cada grupo, creando un resumen a nivel grupo y uniendo los subconjuntos en una base nueva:
summarise(by_genero, promedio = mean(estatura))
#> # A tibble: 2 x 2
#> genero promedio
#> <chr> <dbl>
#> 1 hombre 1.74
#> 2 mujer 1.65 Calcula el retraso promedio por fecha.
Calcula el retraso promedio por fecha.
¿Qué otros resúmenes puedes hacer para explorar el retraso por fecha?
- Algunas funciones útiles con summarise son min(x), median(x), max(x), quantile(x, p), n(), sum(x), sum(x > 1), mean(x > 1), sd(x).
flights$date_only <- as.Date(flights$date)
by_date <- group_by(flights, date_only)
no_miss <- filter(by_date, !is.na(dep))
delays <- summarise(no_miss, mean_delay = mean(dep_delay), n = n())Operador pipeline
En R cuando uno hace varias operaciones es difícil leer y entender el código:
hourly_delay <- filter(summarise(group_by(filter(flights, !is.na(dep_delay)),
date_only, hour), delay = mean(dep_delay), n = n()), n > 10)La dificultad radica en que usualmente los parámetros se asignan después del
nombre de la función usando (). El operador Forward Pipe (%>%) cambia este orden, de manera que un parámetro que precede a la función es enviado ("piped") a la función:x %>% f(y)se vuelvef(x,y),x %>% f(y) %>% g(z)se vuelveg(f(x, y), z)`. Es así que podemos reescribir el código para poder leer las
operaciones que vamos aplicando de izquierda a derecha
y de arriba hacia abajo.
Veamos como cambia el código anterior:
hourly_delay <- flights %>%
filter(!is.na(dep_delay)) %>%
group_by(date_only, hour) %>%
summarise(delay = mean(dep_delay), n = n()) %>%
filter(n > 10)podemos leer %>% como “después”.
 ¿Qué destinos tienen el promedio de retrasos más
alto?
¿Qué destinos tienen el promedio de retrasos más
alto?
¿Qué vuelos (compañía + vuelo) ocurren diario?
En promedio, ¿Cómo varían a lo largo del día los retrasos de vuelos no cancelados? (pista: hour + minute / 60)
Variables por grupo
En ocasiones es conveniente crear variables por grupo, por ejemplo estandarizar dentro de cada grupo z = (x - mean(x)) / sd(x).
Veamos un ejemplo:
planes <- flights %>%
filter(!is.na(arr_delay)) %>%
group_by(plane) %>%
filter(n() > 30)
planes %>%
mutate(z_delay =
(arr_delay - mean(arr_delay)) / sd(arr_delay)) %>%
filter(z_delay > 5)
#> # A tibble: 1,403 x 16
#> # Groups: plane [856]
#> date hour minute dep arr dep_delay arr_delay carrier
#> <dttm> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr>
#> 1 2011-01-28 12:00:00 15 16 1516 1916 351 326 CO
#> 2 2011-01-27 12:00:00 18 22 1822 1945 234 210 CO
#> 3 2011-01-27 12:00:00 21 37 2137 2254 242 219 CO
#> 4 2011-01-27 12:00:00 0 11 11 216 168 137 CO
#> 5 2011-01-27 12:00:00 22 37 2237 153 227 208 CO
#> 6 2011-01-27 12:00:00 21 28 2128 136 231 216 CO
#> 7 2011-01-26 12:00:00 11 46 1146 1633 171 193 CO
#> 8 2011-01-26 12:00:00 9 49 949 1436 144 180 CO
#> 9 2011-01-21 12:00:00 19 11 1911 2352 94 112 CO
#> 10 2011-01-20 12:00:00 6 35 635 807 780 775 CO
#> # ... with 1,393 more rows, and 8 more variables: flight <dbl>,
#> # dest <chr>, plane <chr>, cancelled <dbl>, time <dbl>, dist <dbl>,
#> # date_only <date>, z_delay <dbl>Verbos de dos tablas
¿Cómo mostramos los retrasos de los vuelos en un mapa?
Para responder esta pregunta necesitamos unir la base de datos de vuelos con la de aeropuertos.
location <- airports %>%
select(dest = iata, name = airport, lat, long)
flights %>%
group_by(dest) %>%
filter(!is.na(arr_delay)) %>%
summarise(
arr_delay = mean(arr_delay),
n = n() ) %>%
arrange(desc(arr_delay)) %>%
left_join(location)
#> Joining, by = "dest"
#> # A tibble: 116 x 6
#> dest arr_delay n name lat long
#> <chr> <dbl> <int> <chr> <dbl> <dbl>
#> 1 ANC 26.1 124 Ted Stevens Anchorage International 61.2 -150.
#> 2 CID 17.8 406 Eastern Iowa 41.9 -91.7
#> 3 DSM 16.0 634 Des Moines International 41.5 -93.7
#> 4 SFO 14.9 2800 San Francisco International 37.6 -122.
#> 5 BPT 14.3 3 Southeast Texas Regional 30.0 -94.0
#> 6 GRR 13.7 665 Kent County International 42.9 -85.5
#> 7 DAY 13.7 444 James M Cox Dayton Intl 39.9 -84.2
#> 8 VPS 12.5 864 Eglin Air Force Base 30.5 -86.5
#> 9 ECP 12.4 720 <NA> NA NA
#> 10 SAV 12.3 851 Savannah International 32.1 -81.2
#> # ... with 106 more rowsHay varias maneras de unir dos bases de datos y debemos pensar en el obejtivo:
x <- tibble(name = c("John", "Paul", "George", "Ringo", "Stuart", "Pete"),
instrument = c("guitar", "bass", "guitar", "drums", "bass",
"drums"))
y <- tibble(name = c("John", "Paul", "George", "Ringo", "Brian"),
band = c("TRUE", "TRUE", "TRUE", "TRUE", "FALSE"))
x
#> # A tibble: 6 x 2
#> name instrument
#> <chr> <chr>
#> 1 John guitar
#> 2 Paul bass
#> 3 George guitar
#> 4 Ringo drums
#> 5 Stuart bass
#> 6 Pete drums
y
#> # A tibble: 5 x 2
#> name band
#> <chr> <chr>
#> 1 John TRUE
#> 2 Paul TRUE
#> 3 George TRUE
#> 4 Ringo TRUE
#> 5 Brian FALSE
inner_join(x, y)
#> Joining, by = "name"
#> # A tibble: 4 x 3
#> name instrument band
#> <chr> <chr> <chr>
#> 1 John guitar TRUE
#> 2 Paul bass TRUE
#> 3 George guitar TRUE
#> 4 Ringo drums TRUE
left_join(x, y)
#> Joining, by = "name"
#> # A tibble: 6 x 3
#> name instrument band
#> <chr> <chr> <chr>
#> 1 John guitar TRUE
#> 2 Paul bass TRUE
#> 3 George guitar TRUE
#> 4 Ringo drums TRUE
#> 5 Stuart bass <NA>
#> 6 Pete drums <NA>
semi_join(x, y)
#> Joining, by = "name"
#> # A tibble: 4 x 2
#> name instrument
#> <chr> <chr>
#> 1 John guitar
#> 2 Paul bass
#> 3 George guitar
#> 4 Ringo drums
anti_join(x, y)
#> Joining, by = "name"
#> # A tibble: 2 x 2
#> name instrument
#> <chr> <chr>
#> 1 Stuart bass
#> 2 Pete drumsResumamos lo que observamos arriba:
| Tipo | Acción |
|---|---|
| inner | Incluye únicamente las filas que aparecen tanto en x como en y |
| left | Incluye todas las filas en x y las filas de y que coincidan |
| semi | Incluye las filas de x que coincidan con y |
| anti | Incluye las filas de x que no coinciden con y |
Ahora combinamos datos a nivel hora con condiciones climáticas, ¿cuál es el tipo de unión adecuado?
hourly_delay <- flights %>%
group_by(date_only, hour) %>%
filter(!is.na(dep_delay)) %>%
summarise(
delay = mean(dep_delay),
n = n() ) %>%
filter(n > 10)
delay_weather <- hourly_delay %>% left_join(weather)
#> Joining, by = "hour"
arrange(delay_weather, -delay)
#> # A tibble: 2,091,842 x 17
#> # Groups: date_only [365]
#> date_only hour delay n date temp dew_point humidity
#> <date> <dbl> <dbl> <int> <date> <dbl> <dbl> <dbl>
#> 1 2011-05-12 23 184. 33 2011-01-02 43 28.9 58
#> 2 2011-05-12 23 184. 33 2011-01-03 39 27 62
#> 3 2011-05-12 23 184. 33 2011-01-04 50 45 83
#> 4 2011-05-12 23 184. 33 2011-01-05 62.6 60.8 94
#> 5 2011-05-12 23 184. 33 2011-01-06 53.1 36 52
#> 6 2011-05-12 23 184. 33 2011-01-07 46.9 36 66
#> 7 2011-05-12 23 184. 33 2011-01-08 50 43 77
#> 8 2011-05-12 23 184. 33 2011-01-09 53.1 30 41
#> 9 2011-05-12 23 184. 33 2011-01-10 41 37 86
#> 10 2011-05-12 23 184. 33 2011-01-11 39.9 32 73
#> # ... with 2,091,832 more rows, and 9 more variables: pressure <dbl>,
#> # visibility <dbl>, wind_dir <chr>, wind_dir2 <dbl>, wind_speed <dbl>,
#> # gust_speed <dbl>, precip <dbl>, conditions <chr>, events <chr> ¿Qué condiciones climáticas están asociadas
con retrasos en las salidas de Houston?
¿Qué condiciones climáticas están asociadas
con retrasos en las salidas de Houston?
Explora si los aviones más viejos están asociados a mayores retrasos, responde con una gráfica.
Referencias
Wickham, Hadley. 2011. “The Split-Apply-Combine Strategy for Data Analysis.” Journal of Statistical Software 40 (1):1–29. http://www.jstatsoft.org/v40/i01/.