10.6 Metrópolis
En la sección anterior implementamos el algoritmo de Metrópolis en un caso sencillo: las posiciones eran discretas, en una dimensión y la propuesta era únicamente mover a la izquierda o a la derecha. El algoritmo general aplica para valores continuos, en cualquier número de dimensiones y con distribuciones propuesta más generales. Lo esencial del método no cambia para el caso general:
Metropolis
Tenemos una distribución objetivo \(p(\theta)\) de la cual buscamos generar muestras. Debemos ser capaces de calcular el valor de \(p(\theta)\) para cualquier valor candidato \(\theta\). La distribución objetivo \(p(\theta)\) no tiene que estar normalizada, típicamente \(p(\theta)\) es la distribución posterior de \(\theta\) no normalizada, es decir, es el producto de la verosimilitud y la inicial.
La muestra de la distribución objetivo se genera mediante una caminata aleatoria a través del espacio de parámetros. La caminata inicia en un lugar arbitrario (definido por el usuario). El punto inicial debe ser tal que \(p(\theta)>0\). La caminata avanza en cada tiempo proponiendo un movimiento a una nueva posición y después decidiendo si se acepta o no el valor propuesto. Las distribuciones propuesta pueden tener muchas formas, el objetivo es que la distribución propuesta explore el espacio de parámetros de manera eficiente.
Una vez que tenemos un valor propuesto calculamos: \[p_{mover}=min\bigg( \frac{P(\theta_{propuesta})}{P(\theta_{actual})},1\bigg)\] y aceptamos el valor propuesto con probabilidad \(p_{mover}\)
Y al final obtenemos valores representativos de la distribución objetivo \(\{\theta_1,...,\theta_n\}\)
Es importante recordar que debemos excluir las primeras observaciones pues estas siguen bajo la influencia del valor inicial.
Retomemos el problema de inferencia Bayesiana y veamos como usar el algoritmo de Metrópolis cuando la distribución objetivo es la distribución posterior.
10.6.0.1 Ejemplo: Bernoulli
Retomemos el ejemplo del experimento Bernoulli, iniciamos con una función de distribución que describa nuestro conocimiento inicial y tal que podamos calcular \(p(\theta)\) con facilidad. En este caso elegimos una densidad beta y podemos usar función dbeta de R:
# p(theta) con theta = 0.4, a = 2, b = 2
dbeta(0.4, 2, 2)
#> [1] 1.44
# Definimos la distribución inicial
prior <- function(a = 1, b = 1){
function(theta) dbeta(theta, a, b)
}También necesitamos especificar la función de verosimilitud, en nuestro caso tenemos repeticiones de un experimento Bernoulli por lo que: \[\mathcal{L}(\theta) \propto \theta^{z}(1-\theta)^{N-z}\]
y en R:
# Verosimilitid binomial
likeBern <- function(z, N){
function(theta){
theta ^ z * (1 - theta) ^ (N - z)
}
}Por tanto la distribución posterior \(p(\theta|x)\) es, por la regla de Bayes, proporcional a \(p(x|\theta)p(\theta)\). Usamos este producto como la distribución objetivo en el algoritmo de Metrópolis.
# posterior no normalizada
postRelProb <- function(theta){
mi_like(theta) * mi_prior(theta)
}Implementemos el algoritmo con una inicial \(Beta(1,1)\) (uniforme) y observaciones \(z = \sum{x_i}=11\) y \(N = 14\), es decir lanzamos \(14\) volados de los cuales \(11\) resultan en águila.
# Datos observados
N <- 14
z <- 11
# Defino mi inicial y la verosimilitud
mi_prior <- prior() # inicial uniforme
mi_like <- likeBern(z, N) # verosimilitud de los datos observados
# para cada paso decidimos el movimiento de acuerdo a la siguiente función
caminaAleat <- function(theta){ # theta: valor actual
salto_prop <- rnorm(1, 0, sd = 0.1) # salto propuesto
theta_prop <- theta + salto_prop # theta propuesta
if(theta_prop < 0 | theta_prop > 1){ # si el salto implica salir del dominio
return(theta)
}
u <- runif(1)
p_move <- min(postRelProb(theta_prop) / postRelProb(theta), 1) # prob mover
if(p_move > u){
return(theta_prop) # aceptar valor propuesto
}
else{
return(theta) # rechazar
}
}
set.seed(47405)
pasos <- 6000
camino <- numeric(pasos) # vector que guardará las simulaciones
camino[1] <- 0.1 # valor inicial
# Generamos la caminata aleatoria
for (j in 2:pasos){
camino[j] <- caminaAleat(camino[j - 1])
}
caminata <- data.frame(pasos = 1:pasos, theta = camino)
ggplot(caminata[1:3000, ], aes(x = pasos, y = theta)) +
geom_point(size = 0.8) +
geom_path(alpha = 0.5) +
scale_y_continuous(expression(theta), limits = c(0, 1)) +
scale_x_continuous("Tiempo") +
geom_vline(xintercept = 600, color = "red", alpha = 0.5)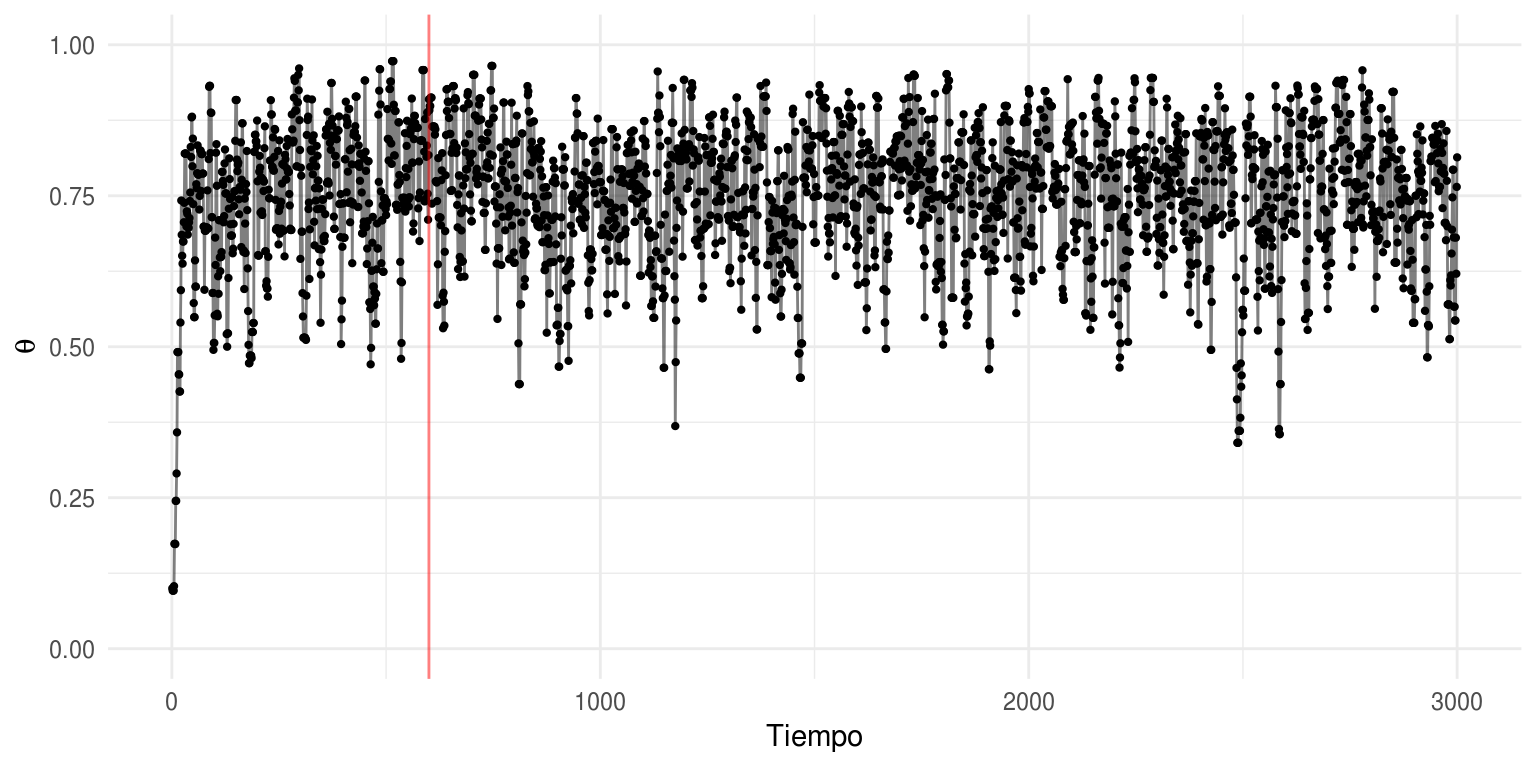
# excluímos las primeras observaciones (etapa de calentamiento)
caminata_f <- filter(caminata, pasos > 600)
ggplot(caminata_f, aes(x = theta)) +
geom_density(adjust = 2, aes(color = "posterior")) +
labs(title = "Distribución posterior",
y = expression(p(theta)),
x = expression(theta)) +
stat_function(fun = mi_prior, aes(color = "inicial")) + # inicial
xlim(0, 1)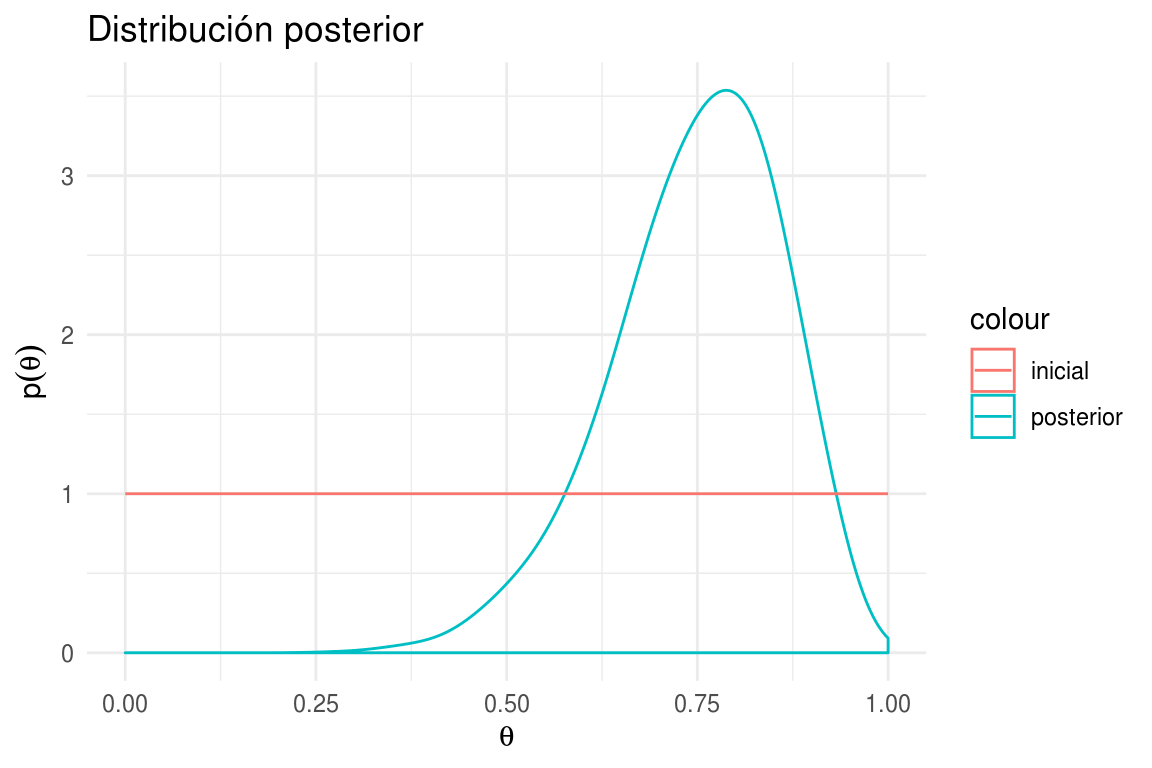
Si la distribución objetivo es muy dispersa y la distribución propuesta muy estrecha, entonces se necesitarán muchos pasos para que la caminata aleatoria cubra la distribución con una muestra representativa.
Por otra parte, si la distribución propuesta es muy dispersa podemos caer en rechazar demasiados valores propuestos. Imaginemos que \(\theta_{actual}\) se ubica en una zona de densidad alta, entonces cuando los valores propuestos están lejos del valor actual se ubicarán en zonas de menor densidad y \(p(\theta_{propuesta})/p(\theta_{actual})\) tenderá a ser chico y el movimiento propuesto será aceptado en pocas ocasiones.
 ¿Qué porcentaje de los valores propuestos son
aceptados? Si cambias la desviación estándar de la distribución propuesta a
\(\sigma = 0.01\) y \(\sigma = 2\), ¿Cómo cambia el porcentaje de aceptación de
valores propuestos? ¿De los \(3\) valores que usamos, qué desviación estándar crees
que sea más conveniente?
¿Qué porcentaje de los valores propuestos son
aceptados? Si cambias la desviación estándar de la distribución propuesta a
\(\sigma = 0.01\) y \(\sigma = 2\), ¿Cómo cambia el porcentaje de aceptación de
valores propuestos? ¿De los \(3\) valores que usamos, qué desviación estándar crees
que sea más conveniente?
De la muestra de valores de \(p(\theta|x)\) obtenidos usando el algoritmo de Metrópolis podemos estimar aspectos de la verdadera distribución \(p(\theta|x)\). Por ejemplo, para resumir la tendencia central es fácil calcular la media y la mediana.
mean(caminata_f$theta)
#> [1] 0.748
sd(caminata_f$theta)
#> [1] 0.109En el caso de predicción:
sims_y <- rbinom(nrow(caminata_f), size = 1, prob = caminata_f$theta)
mean(sims_y) # p(y = 1 | x) probabilidad predictiva
#> [1] 0.75
sd(sims_y)
#> [1] 0.433Inferencia de dos proporciones binomiales
Consideramos la situación en la que nos interesa estudiar dos proporciones \(\theta_1\) y \(\theta_2\) correspondientes a dos grupos. Queremos determinar que debemos creer de estas proporciones tras observar datos provenientes de ambos grupos. Esto es relevante en muchos casos, por ejemplo, en un análisis clínico nos podría interesar evaluar el efecto de una nueva medicina y queremos comoparar la tasa de éxito en el grupo control contra el grupo de tratamiento.
En el marco Bayesiano comenzamos definiendo nuestras creencias iniciales. En este caso nuestras creencias describen combinaciones de parámetros, es decir debemos especificar \(p(\theta_1, \theta_2)\) para todas las combinaciones \(\theta_1, \theta_2\).
Un caso sencillo es asumir que nuestras creencias de \(\theta_1\) son independientes de nuestras creencias de \(\theta_2\). Esto implica:
\[p(\theta_1, \theta_2) = p(\theta_1)p(\theta_2)\] para todo valor de \(\theta_1\) y de \(\theta_2\) y donde \(p(\theta_1)\) y \(p(\theta_2)\) corresponden a las distribuciones marginales. Las creencias de los dos parámetros no tienen porque ser independientes, por ejemplo, puedo creer que dos monedas acuñadas en la misma casa de moneda tienen un sesgo similar, en este caso las manipulaciones matemáticas son más complicadas pero es posible describir estas creencias mediante una distribución bivariada.
Además de las creencias iniciales tenemos datos observados. Suponemos que los lanzamientos dentro de cada grupo son independientes y que los lanzamientos entre los grupos también lo son. Es importante recalcar que siempre suponemos independencia en los datos, sin importar nuestros supuestos de independencia en las creencias.
Para el primer grupo observamos la secuencia \(\{x_{1,1},...,x_{1,N_1}\}\) que contiene \(z_1\) águilas, y en el otro grupo observamos la sucesión \(\{x_{2,1},...,x_{2,N_2}\}\) que contiene \(z_2\) águilas. Es decir, \[z_1 = \sum_{i=1}^{N_1}x_{1,i}\] Por simplicidad denotamos los datos por \(x =\{x_{1,1},...,x_{1,N_1}x_{2,1},...,x_{2,N_2}\}\)
Debido a la independencia de los lanzamientos tenemos:
\[p(x|\theta_1,\theta_2)=\prod_{i=1}^{N_1}p(x_{1,i}|\theta_1,\theta_2)\cdot \prod_{i=1}^{N_2}p(x_{2,i}|\theta_1,\theta_2)\] \[= \theta_1^{z_1}(1-\theta_1)^{N_1-z_1}\cdot \theta_2^{z_2}(1-\theta_2)^{N_2-z_2}\]
Usamos la regla de Bayes para calcular la distribución posterior:
\[p(\theta_1,\theta_2|x)=p(x|\theta_1,\theta_2)p(\theta_1, \theta_2) / p(x)\] \[= \frac{p(x|\theta_1,\theta_2)p(\theta_1, \theta_2)} { \int\int p(x|\theta_1,\theta_2)p(\theta_1, \theta_2)d\theta_1d\theta_2}\]
Distribuciones conjugadas
Siguiendo el caso de la familia Beta-Bernoulli que estudiamos en el caso de una proporción y suponiendo independencia en las creencias, \(p(\theta_1, \theta_2) = p(\theta_1)p(\theta_2)\). Escribimos la densidad inicial como el producto de dos densidades beta, donde \(\theta_1\) se distribuye \(Beta(a_1, b_1)\) y \(\theta_2\) se distribuye \(Beta(a_2, b_2)\).
\[p(\theta_1, \theta_2)=\frac{\theta_1^{a_1-1}(1-\theta)^{b_1-1}} {B(a_1, b_1)}\frac{\cdot \theta_2^{a_2-1}(1-\theta)^{b_2-1}}{B(a_2, b_2)}\]
donde \(B(a, b) = \Gamma(a)\Gamma(b) / \Gamma(a+b)\).
La posterior se escribe:
\[p(\theta_1, \theta_2|x)=\frac{\theta_1^{z_1+a_1-1}(1-\theta)^{b_1-1}\cdot \theta_2^{z_2+a_2-1}(1-\theta)^{b_2-1}}{p(x)\cdot B(a_1, b_1) \cdot B(a_2, b_2)}\] Resumiendo, cuando la inicial es el producto de distribuciones beta independientes, la posterior también es el producto de distribuciones beta independientes.
Veamos las gráficas.
grid <- expand.grid(x = seq(0.01, 1, 0.01), y = seq(0.01, 1, 0.01))
grid_inicial <- grid %>%
mutate(z = round(dbeta(x, 3, 3) * dbeta(y, 3, 3), 1))
binom_2_inicial <- ggplot(grid_inicial, aes(x = x, y = y, z = z)) +
# geom_tile(aes(fill = z)) +
geom_raster(aes(fill = z), show.legend = FALSE) +
geom_contour(colour = "white") +
# stat_contour(binwidth = 0.6, aes(color = ..level..), show.legend = FALSE) +
scale_x_continuous(expression(theta[1]), limits = c(0, 1)) +
scale_y_continuous(expression(theta[2]), limits = c(0, 1)) +
scale_color_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
scale_fill_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
coord_fixed() +
labs(title = "Inicial")
# z_1=5, N_1=7, z_2=2, N_2=7, a_1=a_2=b_1=b_2=3
grid_v <- grid %>%
mutate(z = round(dbeta(x, 6, 3) * dbeta(y, 3, 6), 1))
binom_2_verosimilitud <- ggplot(grid_v, aes(x = x, y = y, z = z)) +
geom_raster(aes(fill = z), show.legend = FALSE) +
geom_contour(colour = "white") +
# stat_contour(binwidth = 0.6, aes(color = ..level..), show.legend = FALSE) +
scale_x_continuous(expression(theta[1]), limits = c(0, 1)) +
scale_y_continuous(expression(theta[2]), limits = c(0, 1)) +
scale_color_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
scale_fill_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
coord_fixed() +
labs(title = "Verosimilitud")
# z_1=5, N_1=7, z_2=2, N_2=7
grid_post <- grid %>%
mutate(z = round(dbeta(x, 8, 5) * dbeta(y, 5, 8), 1))
binom_2_posterior <- ggplot(grid_post, aes(x = x, y = y, z = z)) +
geom_raster(aes(fill = z)) +
geom_contour(colour = "white", show.legend = FALSE) +
# stat_contour(binwidth = 0.6, aes(color = ..level..), show.legend = FALSE) +
scale_x_continuous(expression(theta[1]), limits = c(0, 1)) +
scale_y_continuous(expression(theta[2]), limits = c(0, 1)) +
# scale_color_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
scale_fill_gradient(expression(p(theta[1],theta[2])), limits = c(0, 8.6)) +
coord_fixed() +
labs(title = "Posterior")
grid.arrange(binom_2_inicial, binom_2_verosimilitud, binom_2_posterior,
nrow = 1, widths = c(0.3, 0.3, 0.4))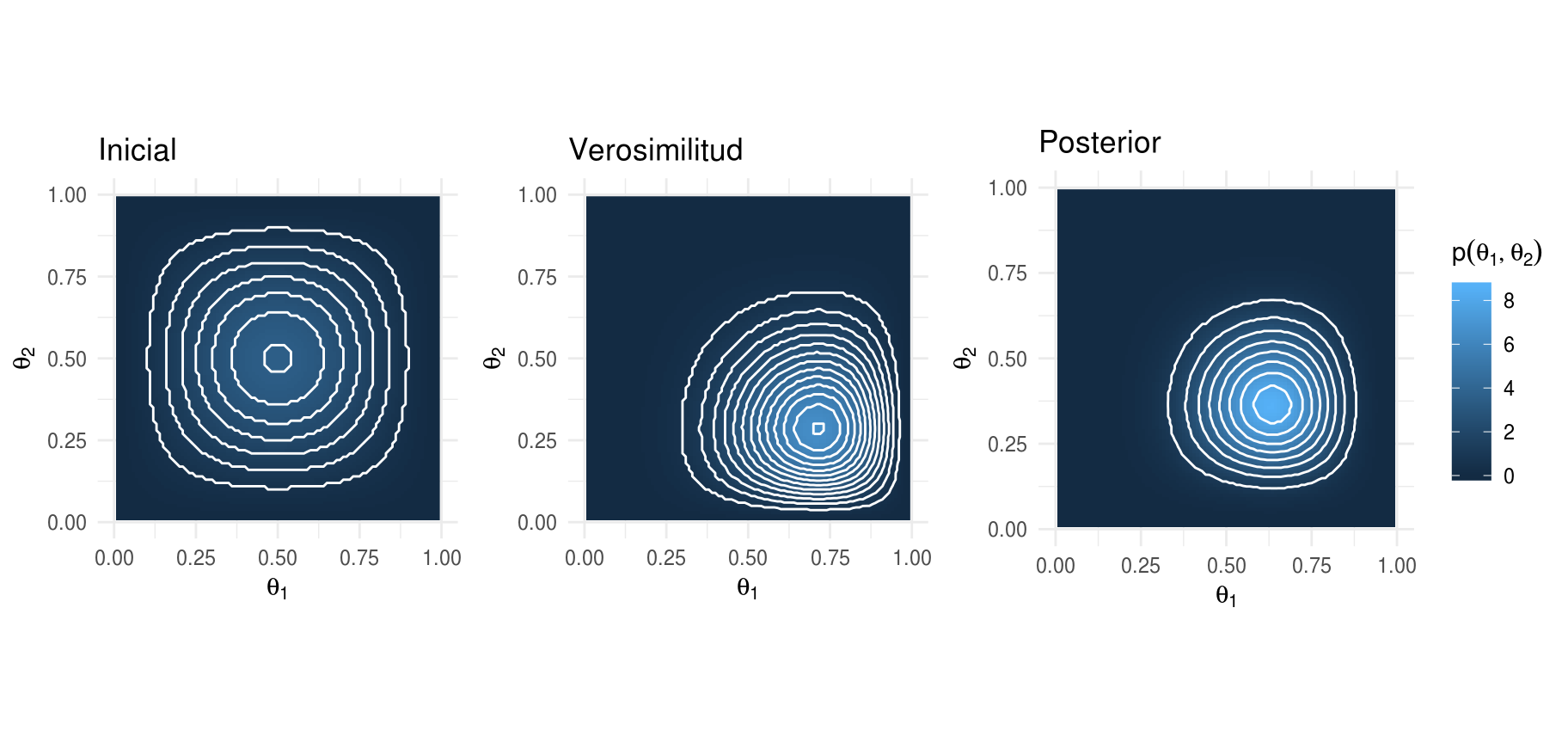
library(plotly)
#>
#> Attaching package: 'plotly'
#> The following object is masked from 'package:ggplot2':
#>
#> last_plot
#> The following object is masked from 'package:stats':
#>
#> filter
#> The following object is masked from 'package:graphics':
#>
#> layout
grid_inicial_pl <- grid_inicial %>% spread(y, z) %>% as.matrix()
pl_inicial <- plot_ly(z = grid_inicial_pl) %>% add_surface(cmin = 0, cmax = 9)
grid_v_pl <- grid_v %>% spread(y, z) %>% as.matrix()
pl_verosimilitud <- plot_ly(z = grid_v_pl) %>% add_surface(cmin = 0, cmax = 9)
grid_post_pl <- grid_post %>% spread(y, z) %>% as.matrix()
pl_post <- plot_ly(z = grid_post_pl) %>% add_surface(cmin = 0, cmax = 9)
pl_inicialpl_verosimilitudpl_postMetrópolis
Al igual que en el caso univariado escribimos la verosimilitud y la distribución inicial:
# Verosimilitid binomial
likeBern2 <- function(z_1, N_1, z_2, N_2){
function(theta){
theta[1] ^ z_1 * (1 - theta[1]) ^ (N_1 - z_1) *
theta[2] ^ z_2 * (1 - theta[2]) ^ (N_2 - z_2)
}
}
prior2 <- function(a_1 = 3, b_1 = 3, a_2 = 3, b_2 = 3){
function(theta) dbeta(theta[1], a_1 , b_1) * dbeta(theta[2], a_2 , b_2)
}
# posterior no normalizada
postRelProb2 <- function(theta){
mi_like(theta) * mi_prior(theta)
}Implementemos el algoritmo con una inicial \(Beta(3,3)\) y observaciones \(z_1=5\), \(N = 7\), \(z_1=2\) y \(N = 7\), es decir lanzamos \(14\) volados \(7\) de cada moneda.
z_1=5; N_1=7; z_2=2; N_2=7; a_1=3; a_2=3; b_1=3; b_2=3
# Datos observados
N = 14
z = 11
# Defino mi inicial y la verosimilitud
mi_prior <- prior2() # inicial uniforme
mi_like <- likeBern2(z_1 = z_1, N_1 = N_1, z_2 = z_2, N_2 = N_2) # verosimilitudPara proponer saltos usaremos una distribución normal bivariada. El movimiento se acepta de manera definitiva si la posterior es más densa que la posición actual y se acepta de manera probabilística si la posición actual es más densa.
# para cada paso decidimos el movimiento de acuerdo a la siguiente función
caminaAleat2 <- function(theta){ # theta: valor actual
salto_prop <- MASS::mvrnorm(n = 1 , mu = rep(0, 2),
Sigma = matrix(c(0.08, 0, 0, 0.08), byrow = TRUE, nrow = 2)) # salto propuesto
theta_prop <- theta + salto_prop # theta propuesta
if(all(theta_prop < 0) | all(theta_prop > 1)){ # salir del dominio
return(theta)
}
u <- runif(1)
p_move = min(postRelProb2(theta_prop) / postRelProb2(theta), 1) # prob mover
if(p_move > u){
return(theta_prop) # aceptar valor propuesto
}
else{
return(theta) # rechazar
}
}
set.seed(47405)
pasos <- 6000
camino <- matrix(0, nrow = pasos, ncol = 2) # vector que guardará las simulaciones
camino[1, ] <- c(0.1, 0.8) # valor inicial
# Generamos la caminata aleatoria
for (j in 2:pasos){
camino[j, ] <- caminaAleat2(camino[j - 1, ])
}
caminata <- data.frame(pasos = 1:pasos, theta_1 = camino[, 1],
theta_2 = camino[, 2])
ggplot(caminata, aes(x = theta_1, y = theta_2)) +
geom_point(size = 0.8) +
geom_path(alpha = 0.3) +
scale_x_continuous(expression(theta[1]), limits = c(0, 1)) +
scale_y_continuous(expression(theta[2]), limits = c(0, 1)) +
coord_fixed()
caminata_m <- caminata %>%
gather(parametro, val, theta_1, theta_2) %>%
arrange(pasos)
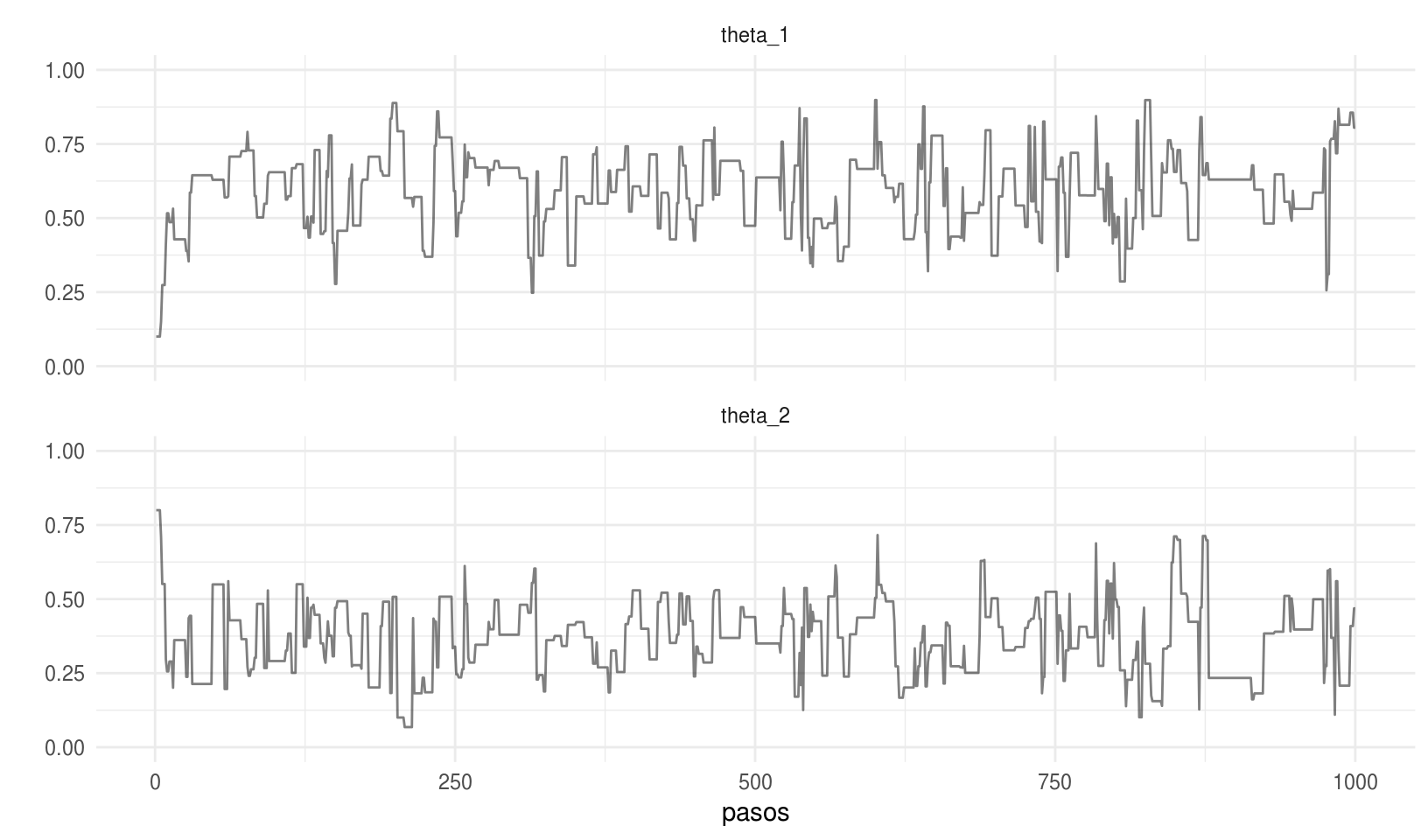
ggplot(caminata_m[1:2000, ], aes (x = pasos, y = val)) +
geom_path(alpha = 0.5) +
facet_wrap(~parametro, ncol = 1) +
scale_y_continuous("", limits = c(0, 1))