4.2 Vectores
En R se puede trabajar con distintas estructuras de datos, algunas son de una sola dimensión y otras permiten más, como indica el diagrama de abajo:
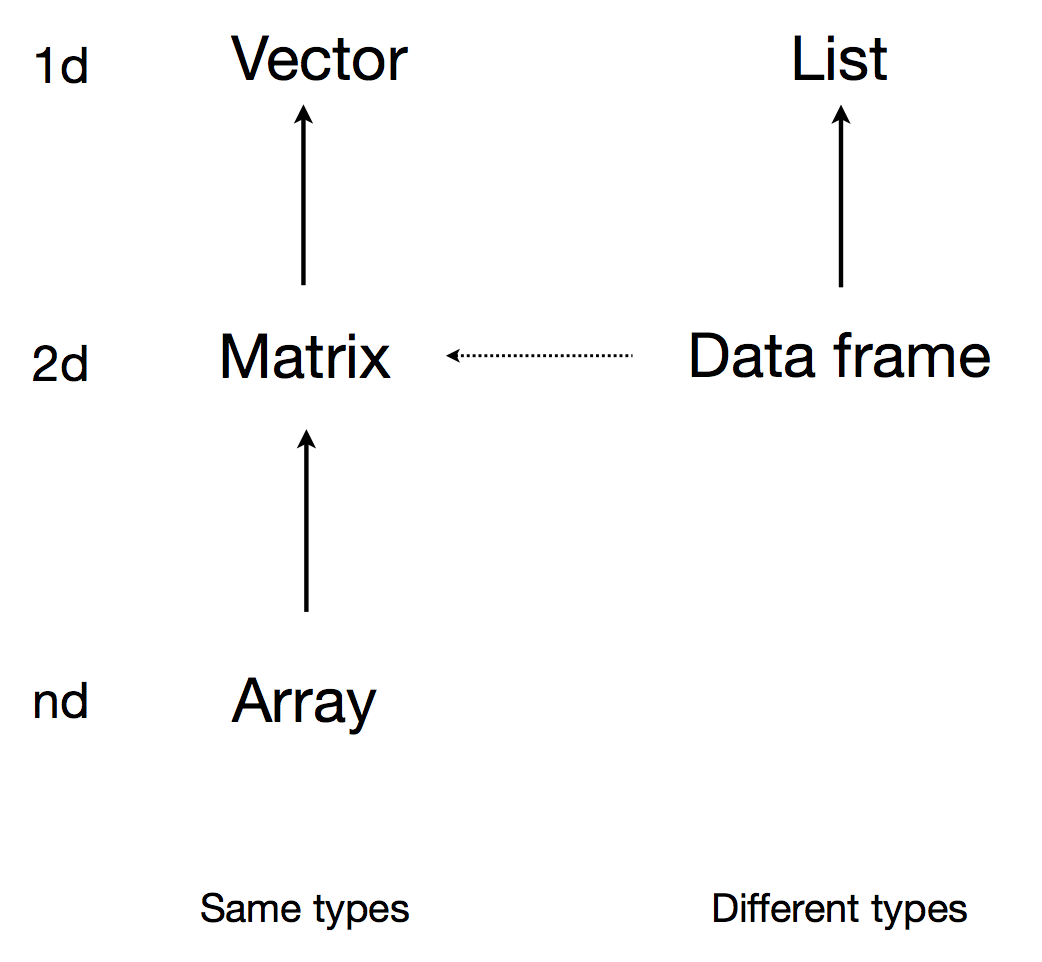
Hasta ahora nos hemos centrado en trabajar con data.frames, y hemos usado
vectores atómicos sin profundizar, en esta sección se explican características
de los vectores, y veremos que son la base de los data.frames.
En R hay dos tipos de vectores, esto es, estructuras de datos de una sola dimensión: los vectores atómicos y las listas.
- Los vectores atómicos pueden ser de 6 tipos: lógico, entero, double, caracter, complejo y raw. Los dos últimos son poco comunes.
Vector atómico de tipo lógico:
a <- c(TRUE, FALSE, FALSE)
a
#> [1] TRUE FALSE FALSENumérico (double):
b <- c(5, 2, 4.1, 7, 9.2)
b
#> [1] 5.0 2.0 4.1 7.0 9.2
b[1]
#> [1] 5
b[2]
#> [1] 2
b[2:4]
#> [1] 2.0 4.1 7.0Las operaciones básicas con vectores atómicos son componente a componente:
c <- b + 10
c
#> [1] 15.0 12.0 14.1 17.0 19.2
d <- sqrt(b)
d
#> [1] 2.236068 1.414214 2.024846 2.645751 3.033150
b + d
#> [1] 7.236068 3.414214 6.124846 9.645751 12.233150
10 * b
#> [1] 50 20 41 70 92
b * d
#> [1] 11.180340 2.828427 8.301867 18.520259 27.904982Y podemos crear secuencias como sigue:
e <- 1:10
e
#> [1] 1 2 3 4 5 6 7 8 9 10
f <- seq(0, 1, 0.25)
f
#> [1] 0.00 0.25 0.50 0.75 1.00Para calcular características de vectores atómicos usamos funciones:
# media del vector
mean(b)
#> [1] 5.46
# suma de sus componentes
sum(b)
#> [1] 27.3
# longitud del vector
length(b)
#> [1] 5Y ejemplo de vector atómico de tipo caracter y funciones:
frutas <- c('manzana', 'manzana', 'pera', 'plátano', 'fresa', "kiwi")
frutas
#> [1] "manzana" "manzana" "pera" "plátano" "fresa" "kiwi"
grep("a", frutas)
#> [1] 1 2 3 4 5
gsub("a", "x", frutas)
#> [1] "mxnzxnx" "mxnzxnx" "perx" "plátxno" "fresx" "kiwi"- Las listas, a diferencia de los vectores atómicos, pueden contener otras listas. Las listas son muy flexibles pues pueden almacenar objetos de cualquier tipo.
x <- list(1:3, "Mila", c(TRUE, FALSE, FALSE), c(2, 5, 3.2))
str(x)
#> List of 4
#> $ : int [1:3] 1 2 3
#> $ : chr "Mila"
#> $ : logi [1:3] TRUE FALSE FALSE
#> $ : num [1:3] 2 5 3.2Las listas son vectores recursivos debido a que pueden almacenar otras listas.
y <- list(list(list(list())))
str(y)
#> List of 1
#> $ :List of 1
#> ..$ :List of 1
#> .. ..$ : list()Para construir subconjuntos a partir de listas usamos [] y [[]]. En el primer
caso siempre obtenemos como resultado una lista:
x_1 <- x[1]
x_1
#> [[1]]
#> [1] 1 2 3
str(x_1)
#> List of 1
#> $ : int [1:3] 1 2 3Y en el caso de [[]] extraemos un componente de la lista, eliminando un nivel
de la jerarquía de la lista.
x_2 <- x[[1]]
x_2
#> [1] 1 2 3
str(x_2)
#> int [1:3] 1 2 3¿Cómo se comparan y, y[1] y y[[1]]?
Propiedades
Todos los vectores (atómicos y listas) tienen las propiedades tipo y longitud,
la función typeof() se usa para determinar el tipo,
=======
Todos los vectores tienen las propiedades tipo y longitud, la función typeof()
se usa para determinar el tipo,
>>>>>>> 27ef834b49e0e8bc1097a6234d308d5406dca20c
typeof(a)
#> [1] "logical"
typeof(b)
#> [1] "double"
typeof(frutas)
#> [1] "character"
typeof(x)
#> [1] "list"y length() la longitud:
length(a)
#> [1] 3
length(frutas)
#> [1] 6
length(x)
#> [1] 4
length(y)
#> [1] 1La flexibilidad de las listas las convierte en estructuras muy útiles y muy comunes, muchas funciones regresan resultados en forma de lista. Incluso podemos ver que un data.frame es una lista de vectores, donde todos los vectores son de la misma longitud.
Adicionalmente, los vectores pueden tener atributo de nombres, que puede usarse para indexar.
names(b) <- c("momo", "mila", "duna", "milu", "moka")
b
#> momo mila duna milu moka
#> 5.0 2.0 4.1 7.0 9.2
b["moka"]
#> moka
#> 9.2names(x) <- c("a", "b", "c", "d")
x
#> $a
#> [1] 1 2 3
#>
#> $b
#> [1] "Mila"
#>
#> $c
#> [1] TRUE FALSE FALSE
#>
#> $d
#> [1] 2.0 5.0 3.2
x$a
#> [1] 1 2 3
x[["c"]]
#> [1] TRUE FALSE FALSE